——基于功能基因/持家基因/靶基因
在自然界的各类环境中,都有微生物的存在,它们在各自的“岗位”上都发挥着或大或小、或多或少的作用。有一些具有特殊功能的微生物,由于其作用的重要性或特殊性而受到人们的广泛关注,这些具有特殊功能的微生物叫做功能微生物,如氨氧化细菌、硫细菌、硝化细菌等。它们在分类学上有可能差异很大,但却具有相同或类似的基因使其能够发挥同样的作用。支配这些功能细菌发挥重要功能的基因被称为功能基因,如amoA、dsrB、nxrA。目前微基生物已收集GeneBank中所有已知功能基因的序列及其对应的种属信息,构建自己的功能基因数据库。 微基生物进行人体健康相关的研究与应用:
微基生物提供与人体健康相关的生信/统计服务:| 功能微生物 | 测序基因 | 特异引物 | 推荐平台 |
| 产甲烷菌 | 16S rRNA gene | Arch519F / Arch915R | MiSeq 2×300 |
| 硫酸盐还原菌SRB | dsrB | dsrF / dsrR | MiSeq 2×300 |
| 硝化细菌AOB | 16S | CTO189f / CTO653r | MiSeq 2×300 |
| 氨氧化细菌AOB | amoA | amoA-1F / amoA-2R | MiSeq 2×300 |
| 氨氧化古菌AOA | amoA | Arch amoA F /Arch amoA R | 454 |
| 硝化细菌NOB | 16S rRNA gene | FGPS1269 / FGPS872 | MiSeq 2×300 |
| 硝化细菌NOB | nxrA | nxrA F / nxrA R | MiSeq 2×300 |
| 厌氧氨氧化菌AMX | 16S rRNA gene | Amx368 / Amx820R | MiSeq 2×300 |
| 反硝化菌 | narG | narG1960f / narG2650r | 454 |
| nirK | F1aCu / R3Cu | MiSeq 2×300 | |
| nirS | cd3aF / R3cd | MiSeq 2×300 | |
| nosZ | nosZF / nosZR | MiSeq 2×300 | |
| 定鞭金藻 | LSU | F / R | MiSeq 2×300 |
| 固碳微生物 | cbbL | 595F / 1387R | 454 |
| cbbM | F / R | MiSeq 2×300 | |
| 固氮微生物 | nifH | PolF / PolR | MiSeq 2×300 |
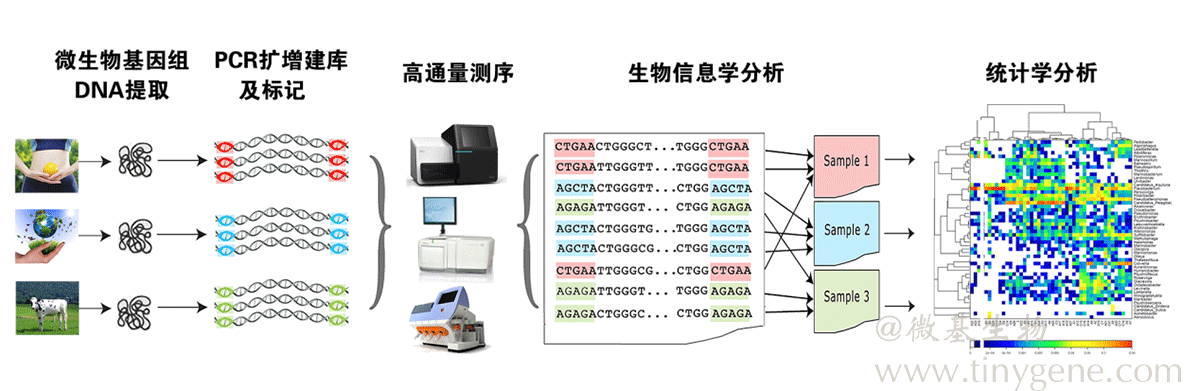
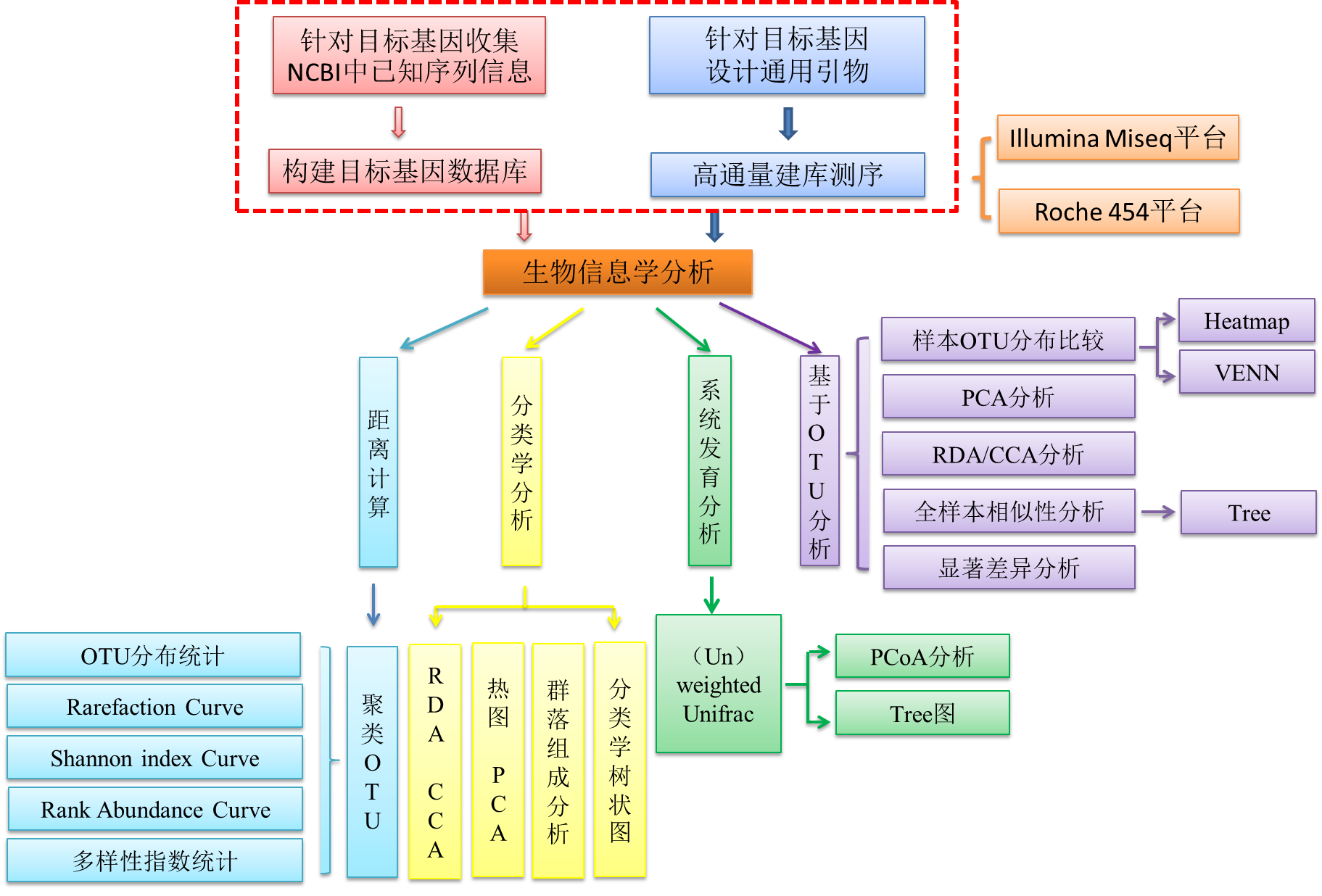
功能基因/持家基因分析的流程图如下:
- 数据库:Silva,GreenGene,RDP
微基生物收集GeneBank中所有已知功能基因的序列及其对应的种属信息,构建自己的功能基因数据库。
目前市面上常用的用于研究环境微生物的高通量测序平台有Illumina,BGI,PacBio和Thermo Ion。其中Illumina平台的测序读长长、测序周期短、通量大,成为常用的测序平台。
微基生物是国内首家采用Illumina MiSeq 2×300 bp平台进行微生物生态研究。在功能基因进行测序分析方面积累了丰富的经验。
- 建库及生信/统计分析
案例分析
标题:Response of denitro bacteria involved in nitrogen removal for treatment of simulated livestock wastewater using a novel bioreactor脱氮细菌在新型生物反应器处理模拟家畜废水脱氮过程中的作用
Ecological Engineering(IF: 3.512)
研究领域:功能微生物
高通量测序平台:Illumina MiSeq
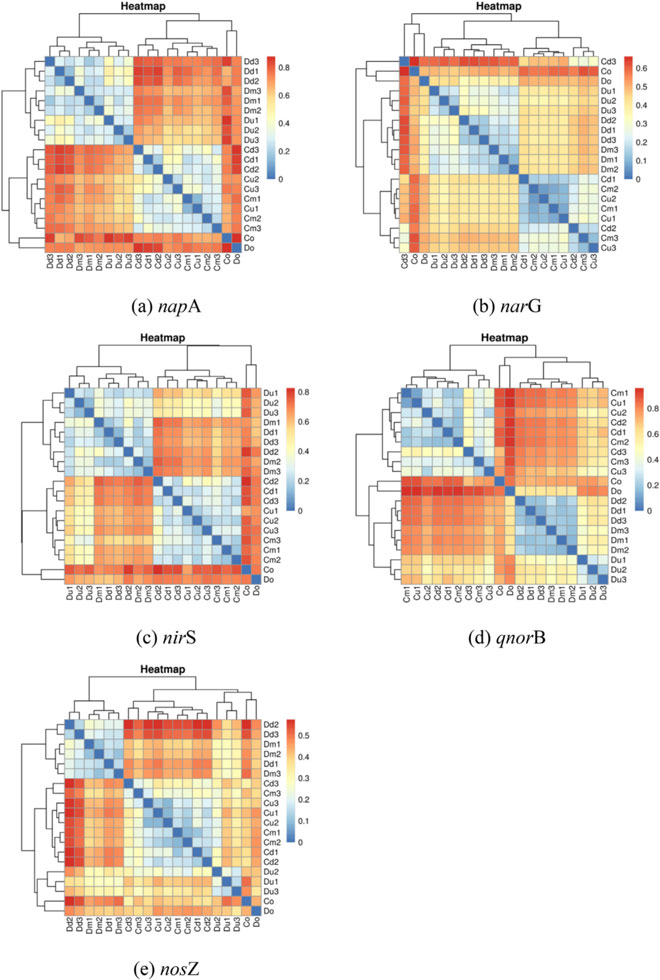
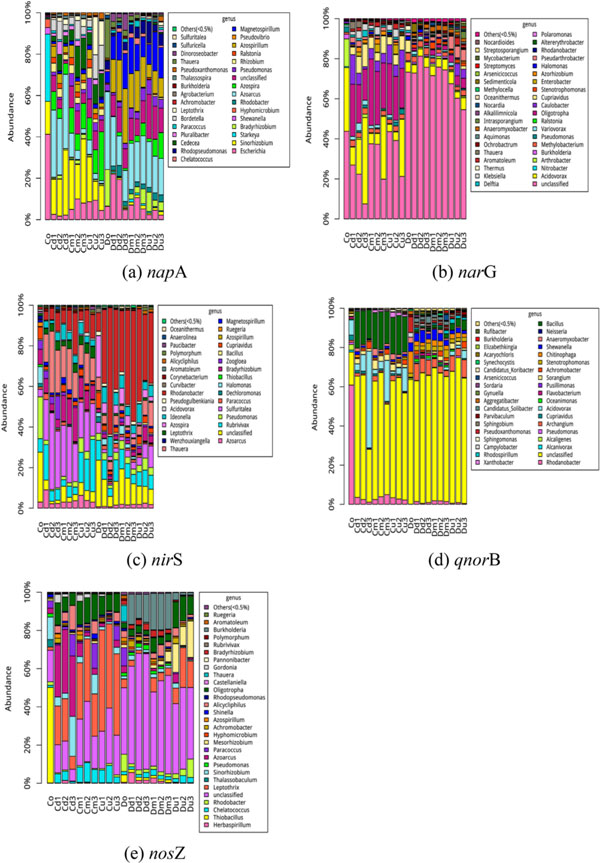
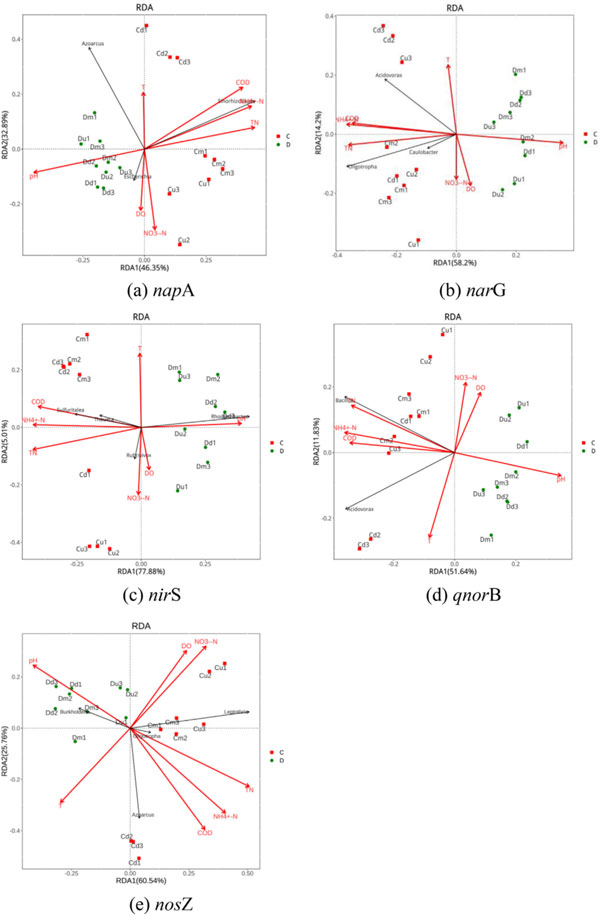
测序区域:napA, narG, nirS, qnorB, and nosZ
主要结果:(1)微生物群落组成的演替反映在CFM组和DAS组之间的显著差异的热图上, 主要体现为中华根瘤菌及固氮菌的不同比例。
(2)环境变量对反硝化群落的存在影响及优势属与氮浓度有一定的相关性
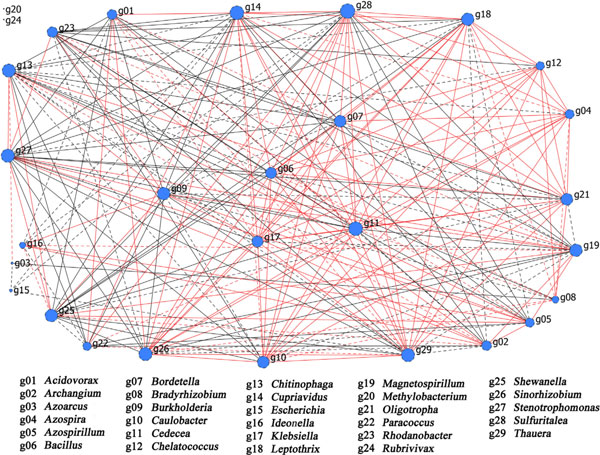
(3)选出29个相对丰度较高的属(每个功能基因选出3 ~ 4个相对丰度最高的属),构建关联网络图。根据它们之间的关联关系,选择相对丰度和适应性较高的菌株,如目标生态位中的Burkholderia, Azoarcus, 和 Rhodanobacter ,加入到细菌接种剂可以实现快速繁殖,并通过自身的酶活性直接改变理化参数。此外,根据系统中的负相关关系,人工增菌可以抑制具有去除化学需氧量和氮的副作用的细菌,从而提高装置的运行效果。
原文链接:https://www.sciencedirect.com/science/article/abs/pii/S0925857420300501
参考文献:Wang L , Xu W , Yu J , et al. Response of denitrobacteria involved in nitrogen removal for treatment of simulated livestock wastewater using a novel bioreactor[J]. Ecological Engineering, 2020, 147:105762.

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用