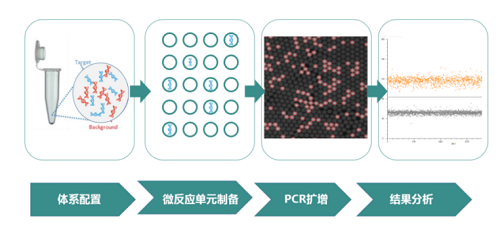
在生物学研究和医学诊断领域,准确检测和定量微生物对于了解疾病机制、制定治疗方案至关重要。然而,当组织样本中存在高宿主DNA残留时,传统的定量方法,如定量聚合酶链式反应(qPCR),常常会受到宿主DNA的严重干扰,导致检测结果出现偏差。而数字滴液PCR(ddPCR)技术的出现,为解决这一难题提供了创新且有效的途径。

ddPCR 检测方法原理
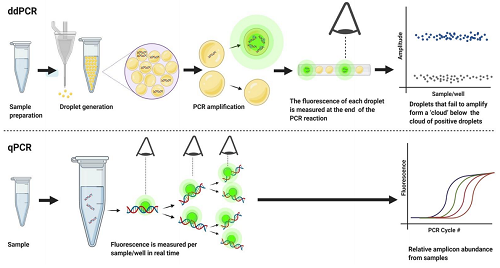
ddPCR是一种基于PCR技术的核酸绝对定量分析方法。它将传统的PCR反应混合物分割成数以万计的纳升级微滴,每个微滴中包含极少量的模板DNA分子以及完整的PCR反应试剂。在PCR扩增过程中,这些微滴相互独立进行反应,互不干扰。最终通过荧光信号检测每个微滴中是否含有目标DNA分子。根据泊松分布原理,通过统计阳性微滴的数量,可以计算出原始样本中目标DNA分子的绝对浓度。
引物和探针的设计在ddPCR检测中起着核心作用,是检测成功的关键环节。合理且精心设计的引物和探针,能够显著提升检测的特异性和灵敏度,进而更精准地识别并检测样本中的目标微生物。
ddPCR特异性菌种 属引物/探针设计
微基生物凭借专业的生物信息学团队和先进的生物信息学软件及工具,严格遵循引物/探针设计原则,为客户量身定制引物和探针。
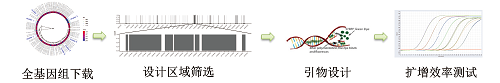
序列分析:对客户提供的目标微生物或基因序列进行全面分析,结合数据库信息,深入研究目标序列特征(GC含量、潜在二级结构、与其他序列的同源性等)。
个性化设计:根据客户实验类型(科研检测、环境监测等)、样本特性(高宿主DNA 残留样本、复杂微生物群落样本等)以及实验目的(定量检测、定性分析等),设计符合特定需求的引物和探针。
多轮优化与验证:设计完成后,借助软件模拟引物和探针在不同条件下与目标序列的结合情况进行多轮虚拟筛选和优化。对设计好的引物和探针进行预实验验证,调整优化,确保其在实际实验中展现出良好的扩增效果和检测性能。
技术支持与服务:为客户提供全方位技术支持,从引物/探针设计咨询、实验方案制定到实验过程指导、结果分析解读等,解答客户在实验过程中遇到的问题。
高宿主DNA残留样本的类型及ddPCR应用
高宿主DNA残留样本常见的样本类型包括:组织样本、粪便样本、口腔拭子样本、血液样本、环境样本以及食品样本等。ddPCR技术凭借其高灵敏度、绝对定量能力和高特异性,提高了检测的准确性和可靠性。

液滴数字PCR (ddPCR)与定量PCR (qPCR)对比
文献分享
1.肿瘤组织中的微生物检测
文献:Accuracy and Clinical Relevance of Intra-Tumoral Fusobacterium nucleatum Detection in Formalin-Fixed Paraffin-Embedded (FFPE) Tissue by Droplet Digital PCR(ddPCR) in Colorectal Cancer.(2022年)
研究背景:Fusobacterium nucleatum(Fn)被认为与结直肠癌的发生和预后密切相关,是一种潜在的预后生物标志物。然而,传统的qPCR方法在检测FFPE组织中的Fn时存在局限性,因此研究者探索了ddPCR技术在检测FFPE组织中Fn的准确性和临床相关性。
研究方法:
1.使用ddPCR技术优化检测条件,包括退火温度和灵敏度评估。
2.在139例CRC FFPE样本中检测Fn DNA,并与qPCR结果进行比较。
3.评估ddPCR检测Fn的准确性,并分析其与临床病理特征和分子特征的相关性。
研究结果:
1.ddPCR检测Fn的检测下限为2.7拷贝/反应,能够检测到20.1%的CRC样本为Fn高表达。
2.与qPCR相比,ddPCR检测Fn的灵敏度更高,但两者结果一致性较低。
3.Fn高表达与CRC的不良预后相关,包括肿瘤位置、分化程度、MSI状态和BRAF突变等。
4.Kaplan-Meier生存分析显示,ddPCR检测到Fn高表达的患者生存率较低,但未达到统计学显著性。
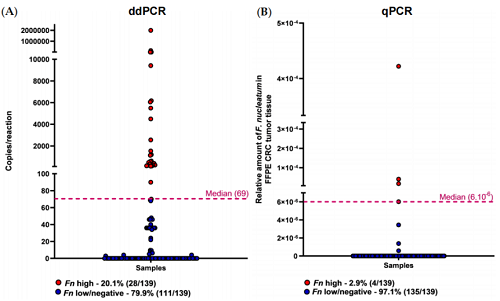
ddPCR和qPCR检测的Fn DNA的量,并基于中位数将样本分为高和低/阴性两组。ddPCR检测到的Fn高样本比例显著高于qPCR。
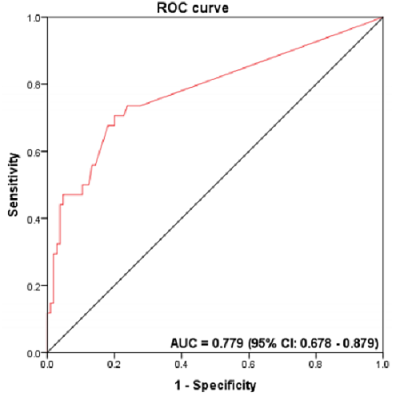
ROC曲线评估ddPCR检测Fn DNA的准确性。ddPCR在检测FFPE样本中的Fn DNA时具有较高的敏感性和特异性。
结论:ddPCR是一种高灵敏度的检测方法,能够准确检测FFPE组织中的Fn DNA,并揭示其在CRC中的临床相关性。ddPCR在常规CRC诊断中具有潜在应用价值。
2.粪便样本中的肠道微生物定量
文献:Detecting and quantifying Veillonella by real-time quantitative PCR and droplet digital PCR.(2024年)
研究背景:Veillonella属细菌是革兰氏阴性机会性病原体,与多种疾病(如牙周炎、炎症性肠病和泌尿系感染)密切相关。研究者设计了一种基于16S rRNA基因序列的引物和探针,通过qPCR和ddPCR技术定量检测粪便样本中的Veillonella丰度。
研究方法:
1.基于16S rRNA基因序列设计引物和探针,进行qPCR和ddPCR检测。
2.使用模拟临床样本测试两种方法的特异性和灵敏度。
3.收集炎症性肠病(IBD)患者的临床样本,验证检测系统的有效性。
研究结果:
1.qPCR的检测灵敏度100拷贝/μL,能够准确检测103到108CFU/mL的Veillonella浓度。
2.ddPCR的检测灵敏度为11.3拷贝/μL,适合检测101到104CFU/mL的低丰度样本。
3.临床样本中,qPCR和ddPCR的检测结果与平板计数法一致,验证了检测系统的准确性。
4.通过ddPCR检测到的Veillonella种类包括V. dispar、V. nakazawae和V. rodentium。
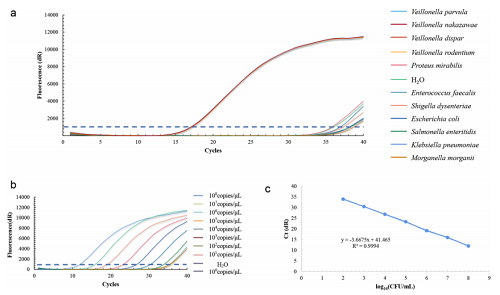
qPCR检测Veillonella的特异性和灵敏度。qPCR的灵敏度100拷贝/μL,适用于检测浓度范围103到108CFU/mL的Veillonella样本。

ddPCR检测Veillonella的特异性和灵敏度。ddPCR的灵敏度11.3拷贝/μL,适用于检测低丰度(101到104CFU/mL)Veillonella样本。
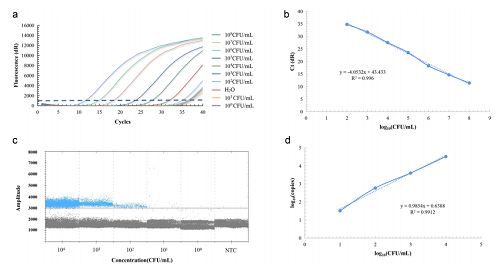
比较qPCR和ddPCR在检测Veillonella时的能力差异。qPCR适用于高浓度样本的检测,而ddPCR更适合低浓度样本的检测。
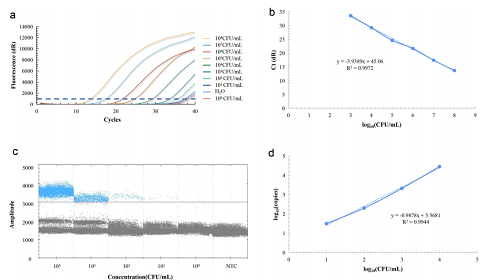
比较qPCR和ddPCR在模拟临床样本(粪便样本)中检测Veillonella的能力。
结论:qPCR和ddPCR技术能够快速、准确地检测Veillonella,ddPCR尤其适用于低丰度样本的检测。
3.血液样本中的病原体检测
文献:Sensitive and rapid identification of pathogens by droplet digital PCR in a cohort of septic patients: a prospective diagnostic study.(2024年)
研究背景:脓毒症是重症监护病房中死亡率较高的疾病之一,早期准确的病原体检测对于改善患者预后至关重要。研究者评估了ddPCR技术在脓毒症患者中快速、灵敏地检测病原体的诊断效能。
研究方法:
1.进行前瞻性诊断研究,纳入89名患者的100例脓毒症发作。
2.使用ddPCR技术检测血液样本中的病原体,并与血培养结果进行比较。
3.评估ddPCR的敏感性、特异性、检测率和周转时间(TAT)。
4.分析经验性抗菌治疗对ddPCR诊断性能的影响,并对ddPCR的定量结果进行动态监测。
研究结果:
1.ddPCR的总体敏感性为75.0%,特异性为69.7%,检测率比血培养高出17%。
2.ddPCR的周转时间为2.5小时,显著快于血培养(中位数47.5小时)。
3.在经验性抗菌治疗组和非经验性抗菌治疗组中,ddPCR的敏感性和特异性无显著差异。
4.ddPCR能够检测到多种抗菌药物耐药基因,且与血培养结果一致。
5.动态监测显示,ddPCR的定量结果与CRP和PCT水平的变化趋势一致,表明其可用于脓毒症的动态监测。

ddPCR检测的实验流程。ddPCR检测的高效性和标准化,适合快速诊断脓毒症患者的病原体。
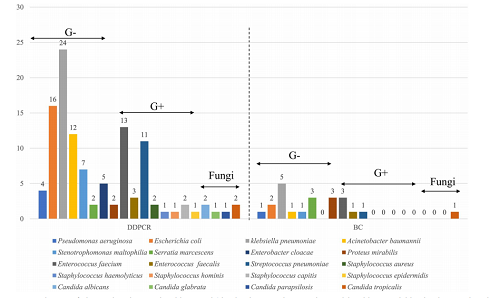
ddPCR和血培养检测到的病原体分布。ddPCR检测病原体种类和数量上优于血培养,能够更全面地识别脓毒症患者的病原体。
结论:与血培养相比,ddPCR在脓毒症患者中具有更高的病原体检测敏感性,能够快速提供病原体和耐药信息,且其定量结果与炎症指标的变化趋势一致,可作为脓毒症患者病原体负荷的动态监测工具。
ddPCR技术凭借其独特的优势,在高宿主DNA残留样本的微生物检测领域展现出巨大的应用潜力。从肿瘤组织到粪便、血液样本,再到复杂的环境样本检测,ddPCR在众多场景下具有实现精准定量检测的潜力,能为微生物检测提供有效的技术支持。
引用文献
1. Datorre, J. G., et al. (2022). Accuracy and Clinical Relevance of Intra-Tumoral Fusobacterium nucleatum Detection in FFPE Tissue by ddPCR in Colorectal Cancer. Diagnostics, 12(1), 114.
2. Ding, Z., et al. (2024). Detecting and Quantifying Veillonella by Real-Time Quantitative PCR and Droplet Digital PCR. Applied Microbiology and Biotechnology, 108, 45.
3. Wu, Z., et al. (2024). Sensitive and Rapid Identification of Pathogens by Droplet Digital PCR in a Cohort of Septic Patients: A Prospective Diagnostic Study. Infectious Diseases, 56(10), 830-841.
4. Kokkoris, V., et al. (2021). Challenges Using Droplet Digital PCR for Environmental Samples. Applied Microbiology, 1, 74-88.

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用



