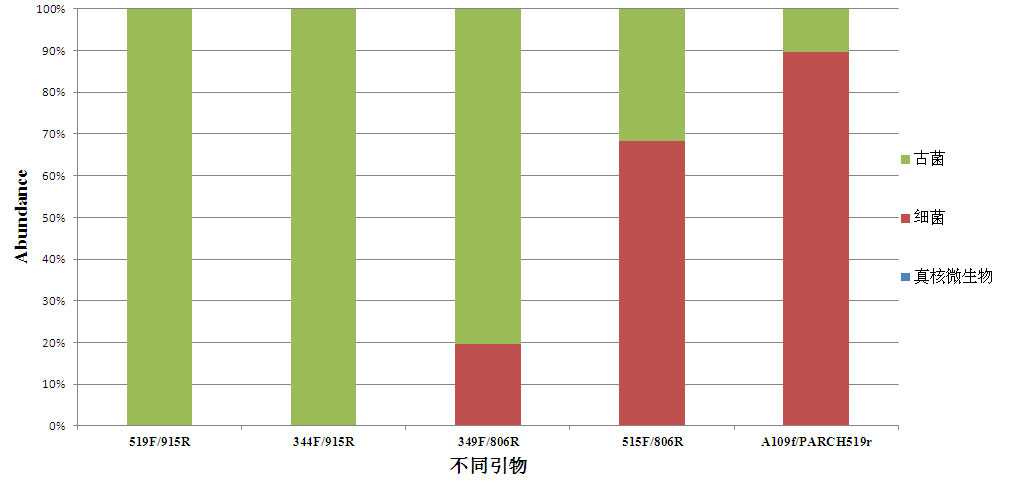
古细菌(archaebacteria)又叫古生菌、古菌,是一类很特殊的细菌,同时具有原核生物和真核生物的某些特征。古细菌多生活在各种极端的生态环境中,它们代表了生命的极限,确定了生物圈的范围。古细菌和真细菌、真核生物一起,构成了生物的三域系统。古细菌分为泉古菌、广古菌、初古菌和纳古菌。具有代表性的古细菌有极端嗜热菌、极端噬盐菌、极端嗜酸菌、产甲烷古菌、嗜热菌、噬盐菌等。 微基生物根据文献中常用的古菌引物,在Silva数据库中搜索,对古细菌测序引物进行了分析比较,结果如下图所示。在所有搜索结果中,引物对519F/915R和344F/915R对于古细菌的特异性较好。其中,引物519F/915R适合Illumina MiSeq 2×300 bp的测序平台,而引物344F/915R则更适合于Roche 454测序平台。
古细菌不同引物对搜索结果
微基生物进行人体健康相关的研究与应用:
微基生物提供与人体健康相关的生信/统计服务: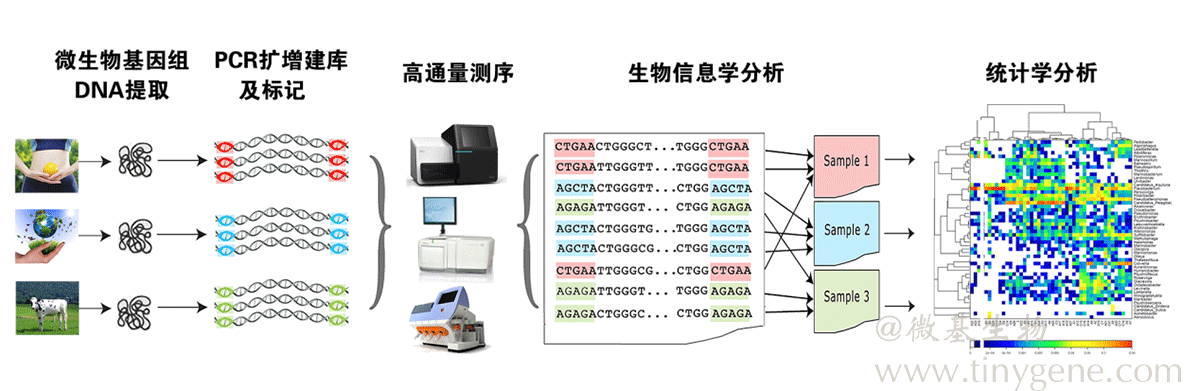
High throughput sequencing flow chart
- 数据库:Silva,GreenGene,RDP
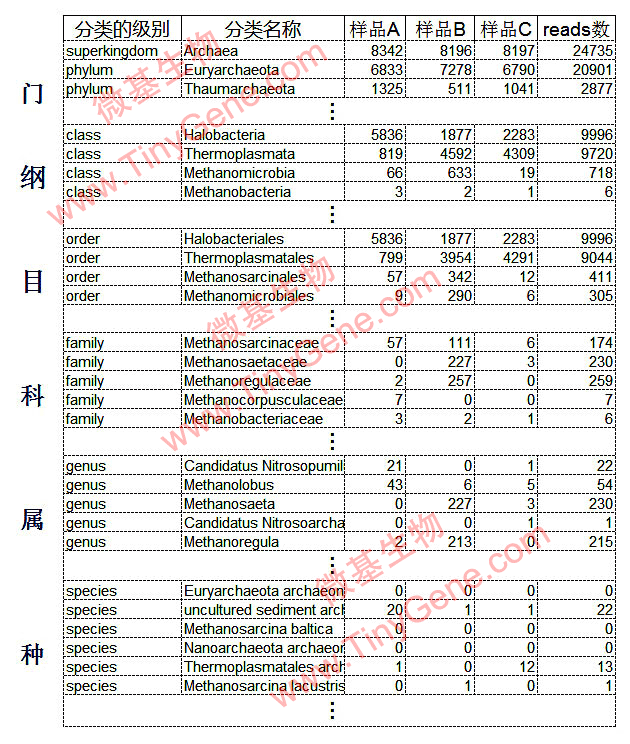
微基生物拥有Silva,GreenGene,RDP三大数据库。其中Silva数据库涵盖了古细菌16S rRNA基因序列及其对应分类信息18797条。
目前市面上常用的用于研究环境微生物的高通量测序平台有Illumina,BGI,PacBio和Thermo Ion。其中Illumina平台的测序读长长、测序周期短、通量大,成为常用的测序平台。
微基生物是国内首家采用Illumina-MiSeq 2×300 bp平台进行微生物生态研究。对古细菌进行测序分析积累了丰富的经验。
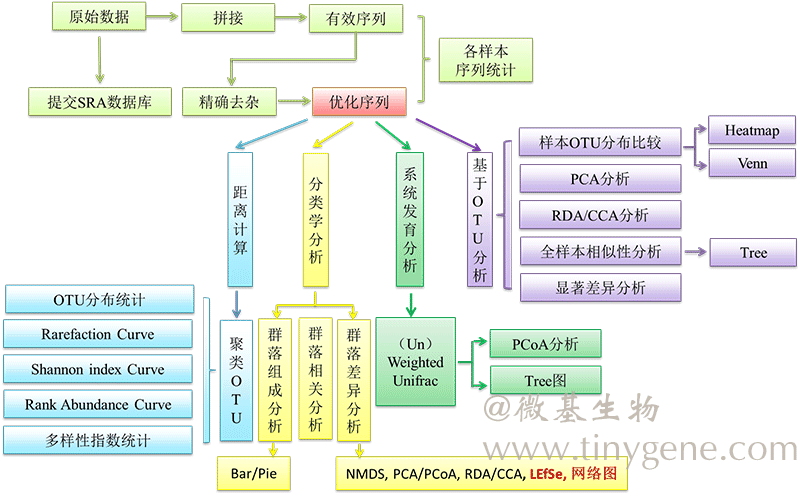
古细菌生物信息分析流程
Bioinfor-statistic analysis
结果展示:
案例分析
标题:在污水处理活性污泥中已知的和未可培养的古细菌种群组成  研究领域:污水处理,活性污泥 分析物种:古细菌 高通量测序平台:Illumina MiSeq 主要结果: (1)12中样品中古细菌的Rarefaction curve
研究领域:污水处理,活性污泥 分析物种:古细菌 高通量测序平台:Illumina MiSeq 主要结果: (1)12中样品中古细菌的Rarefaction curve 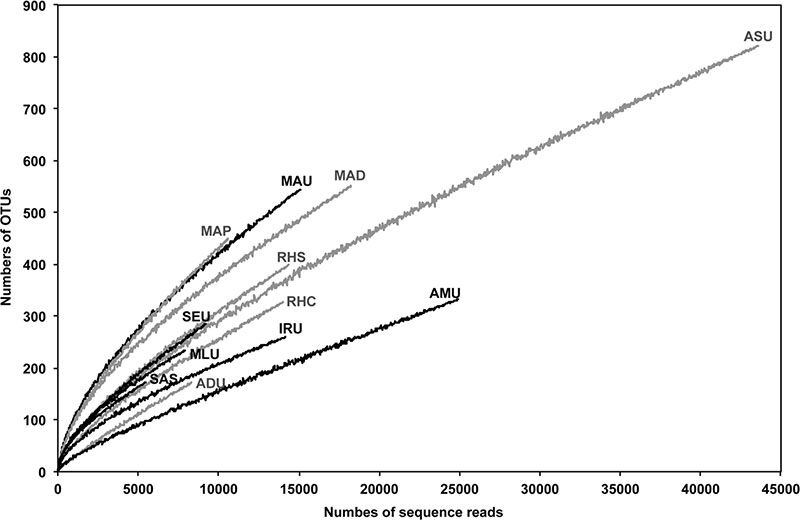 (2)12个污水处理活性污泥样本中古细菌的PCoA分析
(2)12个污水处理活性污泥样本中古细菌的PCoA分析 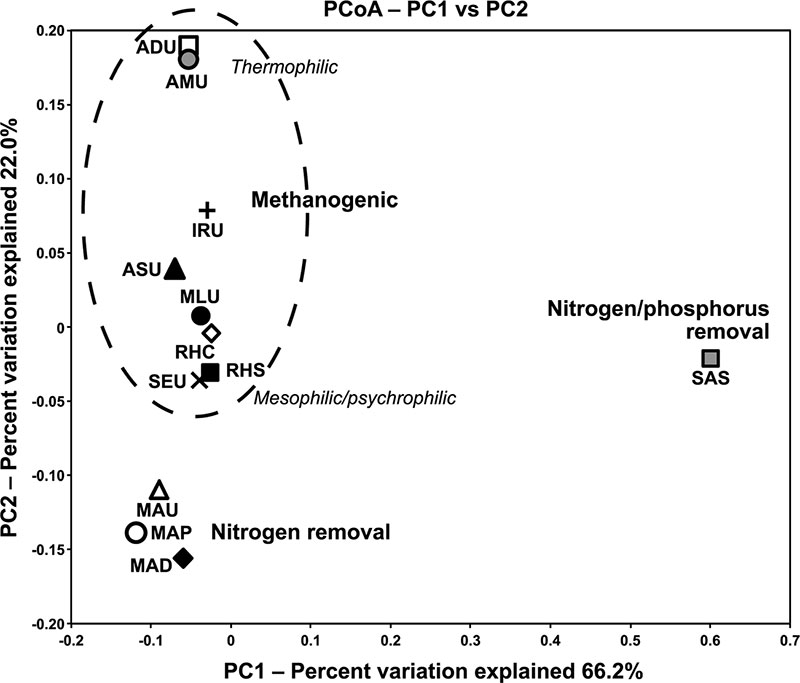 (3)12个活性污泥样品中古细菌的群落组成
(3)12个活性污泥样品中古细菌的群落组成 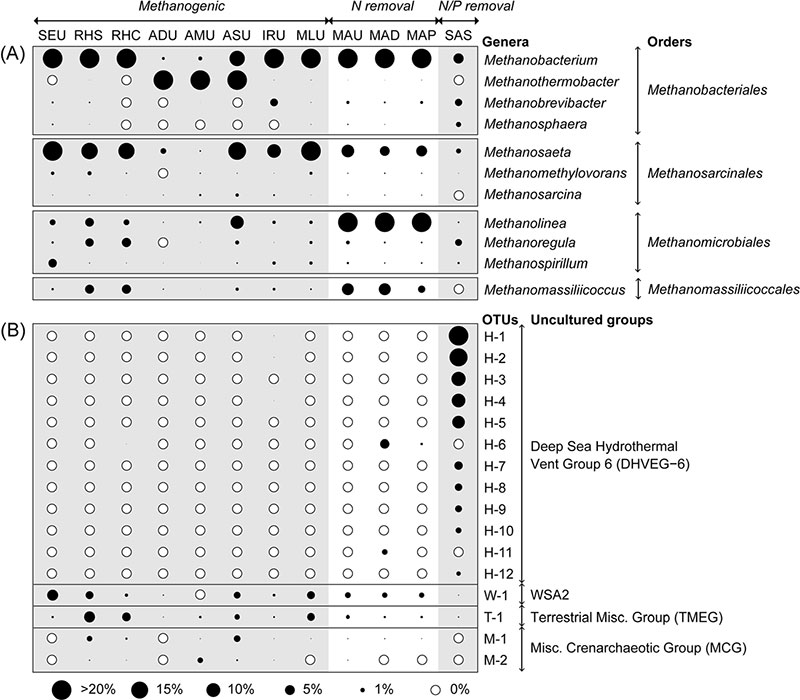
(A)已知的古细菌属;(B)未可培养的古菌
(4)污水处理活性污泥中古细菌的进化树 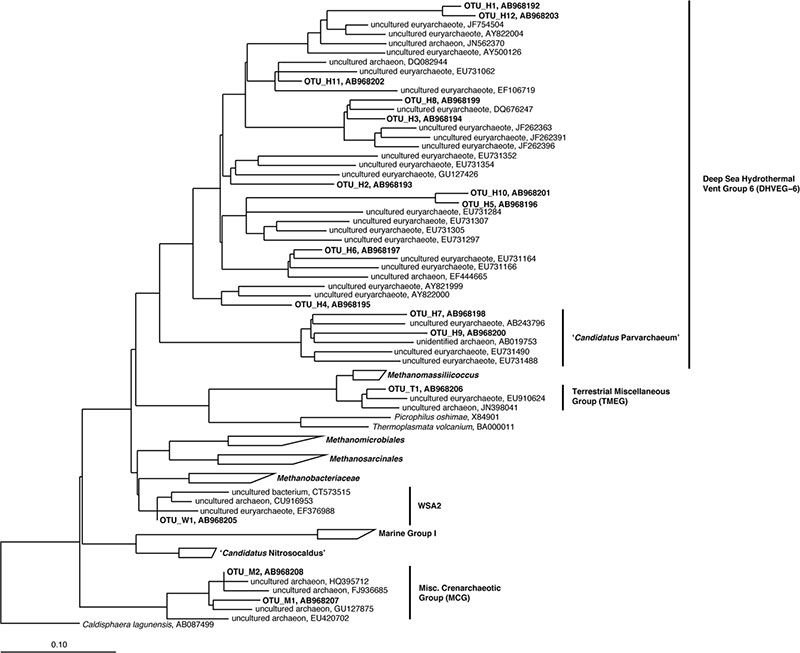 原文链接:http://link.springer.com/article/10.1007/s00248-014-0525-z 参考文献: Kuroda, K., M. Hatamoto, N. Nakahara, K. Abe, M. Takahashi, N. Araki and T. Yamaguchi (2015). “Community composition of known and uncultured archaeal lineages in anaerobic or anoxic wastewater treatment sludge.” Microb Ecol 69(3): 586-596.
原文链接:http://link.springer.com/article/10.1007/s00248-014-0525-z 参考文献: Kuroda, K., M. Hatamoto, N. Nakahara, K. Abe, M. Takahashi, N. Araki and T. Yamaguchi (2015). “Community composition of known and uncultured archaeal lineages in anaerobic or anoxic wastewater treatment sludge.” Microb Ecol 69(3): 586-596.

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用