探针免费设计 专业定制
16S和18S rRNA序列是细菌/古菌和真核生物领域成员的首选靶标,因为它们普遍存在且高度丰富,由高度保守的区域和可变区域组成。FISH技术利用这些基因序列设计特异性的探针,能够精确地定位和识别目标微生物,为微生物群落结构分析、生态功能研究以及疾病诊断等领域提供了强有力的工具。
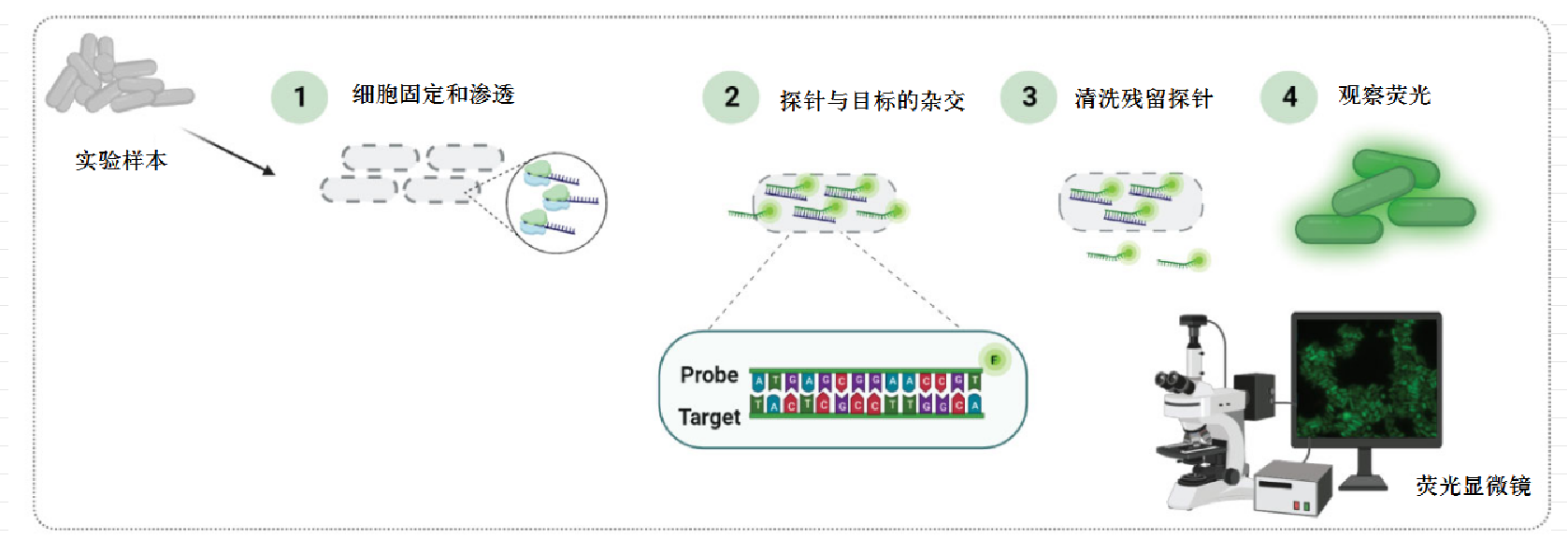
FISH检测的实验流程图:
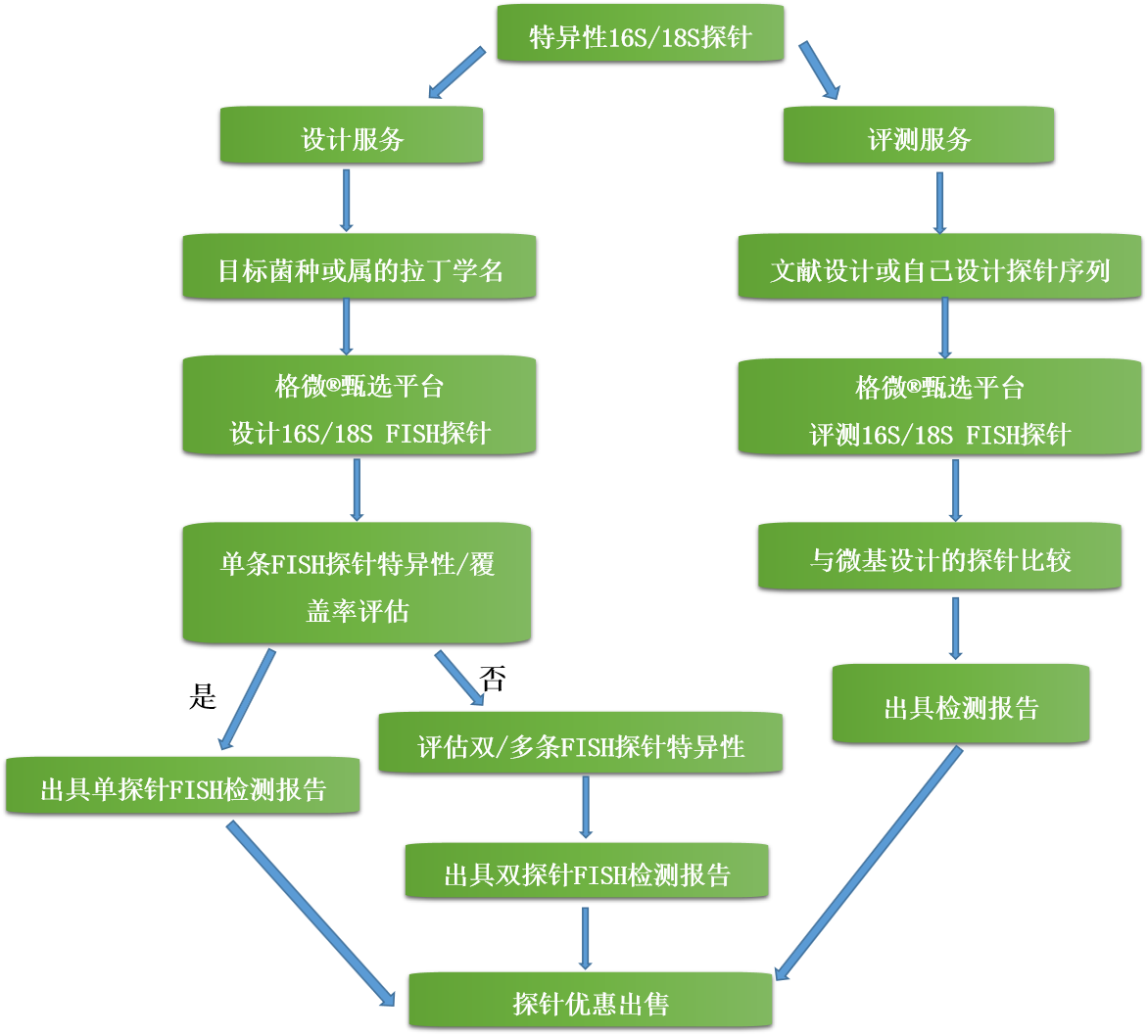
微基生物16S/18S FISH探针设计:
探针设计确实是一个关键步骤,可以影响FISH的性能。微基生物携手格微生物经过几年研发,专为16S/18S FISH探针设计搭建了格微®甄选平台,提供专业、系统性的引物设计与评测。微基生物推出的格微®甄选 菌种特异性16S/18S FISH探针设计、合成及检测服务,将为为科研工作者提供无与伦比的16S/18S FISH探针实验体验。
微基生物已设计完成了100多种微生物“菌种”水平的16S / 18S FISH探针产品,涵盖了多种微生物目标,能够满足不同研究领域的需求。
我们提供个性化产品定制服务,欢迎致电咨询详情:021-50763698。
部分16S / 18S FISH探针商品目录
(注:相关报告可通过以下链接下载查看)
| 1 | G16F1-taxID1716 | 棒状杆菌属 | Corynebacterium | genus | 单探针覆盖率82.05%,双探针覆盖率97.23% |
| 2 | G16F2-taxID1578 | 乳杆菌属 | Lactobacillus | genus | 单探针覆盖率87%,双探针覆盖率97.73% |
| 3 | G16F3-taxID286 | 假单胞菌属 | Pseudomonas | genus | 单探针覆盖率90.10%,双探针覆盖率97.23% |
| 4 | G16F4-taxID13687 | 鞘氨醇单胞菌属 | Sphingomonas | genus | 单探针覆盖率91.99%,双探针覆盖率98.07% |
| 5 | G16F5-taxID1622 | 小鼠乳酸杆菌 | Lactobacillus murinus | species | 单探针覆盖率100% |
| 6 | G16F6-taxID135858 | 链状菌属 | Catenibacterium | genus | 单探针覆盖率92.31%,双探针覆盖率100% |
| 7 | G16F7-taxID2104 | 肺炎支原体 | Mycoplasmoides pneumoniae | species | 单探针覆盖率100% |
| 8 | G16F8-taxID33038 | 瘤胃球菌 | [Ruminococcus] gnavus group | species | 单探针覆盖率91.2%,双探针覆盖率97.6% |
| 9 | G18F9-taxID4929 | 吉氏梅耶酵母 | Meyerozyma guilliermondii | species | 单探针覆盖率9.33%,双探针覆盖率100% |
| 10 | G16F10-taxID469 | 不动杆菌属 | Acinetobacter | genus | 单探针覆盖率91.03%,双探针覆盖率94.64% |
| 11 | G16F11-taxID418240 | 韦氏布劳提菌 | Blautia wexlerae | species | 单探针覆盖率100% |
| 12 | G16F12-taxID1763 | 分枝杆菌属 | Mycobacterium | genus | 单探针覆盖率89.12%,双探针覆盖率97.14% |
| 13 | G18F13-taxID5477 | 白念珠菌 | [Candida] boidinii | species | 单探针覆盖率100% |
| 14 | G16F14-taxID33053 | 粉红奈瑟菌 | Neisseria perflava | species | 单探针覆盖率91.67%,双探针覆盖率100% |
| 15 | G16F15-taxID1396 | 蜡样芽孢杆菌 | Bacillus cereus | species | 单探针覆盖率91.31%,双探针覆盖率97.24% |
| 16 | G16F16-taxID1505 | 索氏梭菌 | Paeniclostridium sordellii | species | 单探针覆盖率92.59%,双探针覆盖率92.30% |
| 17 | G16F17-taxID851 | 具核梭杆菌 | Fusobacterium nucleatum | species | 单探针覆盖率89.05%,双探针覆盖率100% |
| 18 | G16F18-taxID1694 | 假长双歧杆菌 | Bifidobacterium pseudolongum | species | 单探针覆盖率100% |
| 19 | G16F19-taxID38313 | 海藻希瓦氏菌 | Shewanella algae | species | 单探针覆盖率90.70%,双探针覆盖率97.67% |
| 20 | G16F20-taxID24 | 腐败希瓦氏菌 | Shewanella putrefaciens | species | 单探针覆盖率87.5%,双探针覆盖率92.19% |
| 21 | G16F21-taxID40215 | 嗜胶菌属菌俊尼氏菌 | Acinetobacter junii | species | 单探针覆盖率85.25%,双探针覆盖率93.44% |
| 22 | G16F22-taxID1313 | 肺炎链球菌 | Streptococcus pneumoniae | species | 单探针覆盖率89.61%,双探针覆盖率92.58% |
| 23 | G16F23-taxID492670 | 贝莱斯芽孢杆菌 | Bacillus velezensis | species | 单探针覆盖率90.07%,双探针覆盖率97.88% |
| 24 | G16F24-taxID58231 | 中间螺旋体菌 | Treponema medium | species | 单探针覆盖率100% |
| 25 | G16F25-taxID301302 | 粪罗斯氏菌 | Roseburia faecis | species | 单探针覆盖率100% |
| 26 | G16F26-taxID820 | 单形拟杆菌 | Bacteroides uniformis | species | 单探针覆盖率90%,双探针覆盖率100% |
| 27 | G16F27-taxID28135 | 口炎普雷沃菌 | Prevotella oris | species | 单探针覆盖率100% |
| 28 | G16F28-taxID244366 | 栖异地克雷伯氏菌 | Klebsiella variicola | species | 单探针覆盖率94.07%,双探针覆盖率97.04% |
| 29 | G16F29-taxID489 | 多糖奈瑟菌 | Neisseria polysaccharea | species | 单探针覆盖率85.71%,双探针覆盖率100% |
| 30 | G16F30-taxID90371 | 肠沙门氏菌肠亚种鼠伤寒血清型 | Salmonella enterica subsp. enterica serovar Typhimurium | no rank | 单探针覆盖率96.28%,双探针覆盖率97.02% |
| 31 | G16F31-taxID316435 | 大肠杆菌Nissle 1917 | Escherichia coli Nissle 1917 | strain | 单探针覆盖率88.24%,双探针覆盖率100% |
| 32 | G16F32-taxID453840 | 潮栖阿利霍夫莱菌 | Aliihoeflea aestuarii | species | 单探针覆盖率100% |
| 33 | G16F33-taxID1747 | 痤疮丙酸杆菌 | Cutibacterium acnes | species | 单探针覆盖率88.61%,双探针覆盖率96.20% |
| 34 | G16F34-taxID1679 | 长双歧杆菌长亚种 | Bifidobacterium longum subsp.longum Reuter | subspecies | 单探针覆盖率100% |
| 35 | G16F35-taxID216816 | 长双歧杆菌 | Bifidobacterium longum | species | 单探针覆盖率93.88%,双探针覆盖率96.94% |
| 36 | G16F36-taxID1522- | 无害梭菌群 | [Clostridium] innocuum group | species | 单探针覆盖率84.82%,双探针覆盖率94.64% |
| 37 | G16F37-taxID1522 | 无害梭菌 | [Clostridium] innocuum | species | 单探针覆盖率85.71%,双探针覆盖率100% |
| 38 | G16F38-taxID1862672 | 纽约杜波西拉菌 | Dubosiella newyorkensis | species | 单探针覆盖率100% |
| 39 | G16F39-taxID46124 | 颗粒链球菌 | Granulicatella adiacens | species | 单探针覆盖率100% |
| 40 | G16F40-taxID210 | 幽门螺杆菌 | Helicobacter pylori | species | 单探针覆盖率97.34%,双探针覆盖率99.11% |
| 41 | G16F41-taxID484 | 黄染奈瑟菌 | Neisseria flavescens | species | 单探针覆盖率85.71%,双探针覆盖率100% |
| 42 | G16F42-taxID40324 | 嗜麦芽寡养单胞菌 | Stenotrophomonas maltophilia | species | 单探针覆盖率86.82%,双探针覆盖率96.90% |
| 43 | G16F43-taxID239935 | 嗜黏蛋白阿克曼菌 | Akkermansia muciniphila | species | 单探针覆盖率100% |
| 44 | G16F44-taxID2100 | 猪鼻支原体 | Mycoplasma hyorhinis | species | 单探针覆盖率100% |
| 45 | G16F45-taxID573 | 肺炎克雷伯菌 | Klebsiella pneumoniae | species | 单探针覆盖率95.64%,双探针覆盖率98.88% |
| 46 | G16F46-taxID1825023 | 硝态纤细菌候选种 | Candidatus Nitrosotenuis | genus | 单探针覆盖率88.24%,双探针覆盖率97.06% |
| 47 | G16F47-taxID963 | 草生螺杆菌属 | Herbaspirillum | genus | 单探针覆盖率91.08%,双探针覆盖率96.82% |
| 48 | G16F48-taxID2301481 | 未培养的Muribaculaceae细菌 | uncultured Muribaculaceae bacterium | species | 单探针覆盖率100% |
| 49 | G16F49-taxID1678 | 双歧杆菌属 | Bifidobacterium | genus | 单探针覆盖率91.10%,双探针覆盖率97.88% |
| 50 | G16F50-taxID216851 | 粪便菌属 | Faecalibacterium | genus | 单探针覆盖率90.51%,双探针覆盖率98.41% |
| 51 | G16F51-taxID2767887 | 丽珠乳酸菌属 | Ligilactobacillus | genus | 单探针覆盖率94.44%,双探针覆盖率97.34% |
| 52 | G16F52-taxID84111 | 埃氏菌属 | Eggerthella | genus | 单探针覆盖率83.33%,双探针覆盖率86.11% |
| 53 | G16F53-taxID459786 | 摆动菌属 | Oscillibacter | genus | 单探针覆盖率93.38%,双探针覆盖率97.63% |
| 54 | G16F54-taxID470 | 鲍氏不动杆菌 | Acinetobacter baumannii | species | 单探针覆盖率84.51%,双探针覆盖率98.21% |
| 55 | G16F55-taxID562 | 大肠埃希氏菌 | Escherichia coli | species | 单探针覆盖率93.70%,双探针覆盖率99.57% |
| 56 | G16F56-taxID727 | 流感嗜血杆菌 | Haemophilus influenzae | species | 单探针覆盖率88.10%,双探针覆盖率98.73% |
| 57 | G16F57-taxID287 | 铜绿假单胞菌 | Pseudomonas aeruginosa | species | 单探针覆盖率91.88%,双探针覆盖率97.15% |
| 58 | G16F58-taxID1280 | 金黄色葡萄球菌 | Staphylococcus aureus | species | 单探针覆盖率90.64%,双探针覆盖率97.94% |
| 59 | G16F59-taxID1532 | 圆形布劳提氏菌 | Blautia coccoides | species | 单探针覆盖率100% |
| 60 | G16F60-taxID871665 | 类球布劳特氏菌 | Blautia faecis | species | 单探针覆盖率100% |
| 61 | G16F61-taxID536633 | 葡萄糖布劳提氏菌 | Blautia glucerasea | species | 单探针覆盖率100% |
| 62 | G16F62-taxID53443 | 产氢布劳提氏菌 | Blautia hydrogenotrophica | species | 单探针覆盖率100% |
| 63 | G16F63-taxID89014 | 鲁氏布劳提氏菌 | Blautia luti | species | 单探针覆盖率100% |
| 64 | G16F64-taxID40520 | 欧氏布劳提氏菌 | Blautia obeum | species | 单探针覆盖率83.33%,双探针覆盖率100% |
| 65 | G16F65-taxID33035 | 产气布劳提氏菌 | Blautia producta | species | 单探针覆盖率100% |
| 66 | G16F66-taxID871664 | 劳特氏菌 | Blautia stercoris | species | 单探针覆盖率100% |
| 67 | G16F67-taxID42684 | 丘陵链霉菌 | Streptomyces collinus | species | 单探针覆盖率89.47% |
| 68 | G16F68-taxID28125 | 双路普雷沃菌 | Prevotella bivia | species | 单探针覆盖率88.89%,双探针覆盖率100% |
| 69 | G16F69-taxID329854 | 肠道拟杆菌 | Bacteroides intestinalis | species | 单探针覆盖率100% |
| 70 | G16F70-taxID43768 | 马氏棒状杆菌 | Corynebacterium matruchoti | species | 单探针覆盖率100% |
| 71 | G16F71-taxID564 | 弗格森埃希菌 | Escherichia fergusonii | species | 单探针覆盖率97.14%,双探针覆盖率100% |
| 72 | G16F72-taxID1485 | 梭菌属 | Clostridium | genus | 单探针覆盖率100% |
| 73 | G16F73-taxID1926283 | 爱德华梭菌 | Lachnoclostridium edouardi | species | 单探针覆盖率100% |
| 74 | G16F74-taxID496 | 猕猴奈瑟菌 | Neisseria macacae | species | 单探针覆盖率100% |
| 75 | G16F75-taxID158822 | 尼特氏菌M006株 | Cedecea neteri strain M006 | species | 单探针覆盖率100% |
| 76 | G16F76-taxID230143 | 斯卡多维亚菌 | Scardovia wiggsiae DSM22547 | species | 单探针覆盖率100% |
| 77 | G16F77-taxID3068309 | 瘤胃球菌科UCG-005 | Ruminococcaceae bacterium UCG-005 | species | 单探针覆盖率81.74%,双探针覆盖率91.82% |
| 78 | G16F78-taxID1598 | 雷特氏乳酸菌CICC 6132株 | Lactobacillus reuteri CICC 6132 | strain | 单探针覆盖率100% |
| 79 | G16F79-taxID29546 | 胞内劳森菌 | Lawsonia intracellularis | species | 单探针覆盖率100% |
| 80 | G16F80-taxID837 | 牙龈卟啉单胞菌 | Porphyromonas gingivalis | species | 单探针覆盖率98.97% |
| 81 | G16F81-taxID2702 | 阴道加德纳菌 | Gardnerella vaginalis | species | 单探针覆盖率94.44%,双探针覆盖率100% |
| 82 | G16F82-taxID147802 | 惰性乳酸杆菌 | Lactobacillus iners | species | 单探针覆盖率100% |
| 83 | G16F83-taxID528 | 苍白杆菌属 | Ochrobactrum | genus | 单探针覆盖率92.07%,双探针覆盖率95.47% |
| 84 | G16F84-taxID39486 | 长链多尔氏菌 | Dorea formicigenerans | species | 单探针覆盖率100% |
| 85 | G16F85-taxID28139 | 小梭文肯菌 | Rikenella microfusus | species | 单探针覆盖率100% |
| 86 | G16F86-taxID43675 | 粘滑罗氏菌 | Rothia mucilaginosa | species | 单探针覆盖率94.12%,双探针覆盖率100% |
| 87 | G16F87-taxID35832 | 双叶菌属 | Bilophila | genus | 单探针覆盖率91.67%,双探针覆盖率95.83% |
| 88 | G16F88-taxID872 | 脱硫弧菌属 | Desulfovibrio | genus | 单探针覆盖率81.05%,双探针覆盖率97.89% |
| 89 | G16F89-taxID292800 | 普劳氏黄酮分解菌 | Flavonifractor plautii | species | 单探针覆盖率85.71%,双探针覆盖率100% |
| 90 | G16F90-taxID92488 | 内生潘托菌 | Pantoea endophytica | species | 单探针覆盖率100% |
| 91 | G16F91-taxID435590 | 普通类杆菌 ATCC 8482 | Bacteroides vulgatus ATCC 8482 | strain | 单探针覆盖率85.71%,双探针覆盖率100% |
| 92 | G16F92-taxID1673609 | 阿尔本斯不动杆菌 | Acinetobacter albensis | species | 单探针覆盖率100% |
| 93 | G16F93-taxID40214 | 约氏不动杆菌 | Acinetobacter johnsonii | species | 单探针覆盖率88.71%,双探针覆盖率93.55% |
| 94 | G16F94-taxID13690 | 斯菲金戈属 | Sphingobium yanoikuyae | species | 单探针覆盖率96.3% |
| 95 | G16F95-taxID360807 | 菊粉利用罗斯伯里菌 | Roseburia inulinivorans | species | 单探针覆盖率100% |
| 96 | G16F96-taxID85831 | 产酸梭菌 | Bacteroides acidifaciens | species | 单探针覆盖率100% |
| 97 | G16F97-taxID1351 | 粪肠球菌 | Enterococcus faecalis | species | 单探针覆盖率88.52%,双探针覆盖率95.47% |
| 98 | G16F98-taxID40520 | 肥胖拟杆菌 | Blautia obeum | species | 单探针覆盖率83.33%,双探针覆盖率100% |
| 99 | G16F99-taxID1827 | 红球菌属 | Rhodococcus | genus | 单探针覆盖率91.48%,双探针覆盖率98.43% |
| 100 | G16F100-taxID964 | 草根螺菌 | Herbaspirillum seropedicae | species | 单探针覆盖率95.24%,双探针覆盖率100% |
| 101 | G16F101-taxID47678 | 一种肠道拟杆菌 | Bacteroides caccae | species | 单探针覆盖率100% |
| 102 | G16F102-taxID654 | 韦罗气单胞菌 | Aeromonas veronii | species | 单探针覆盖率97.09%,双探针覆盖率98.84% |
| 103 | G16F103-taxID853 | 普氏粪肠杆菌 | Faecalibacterium prausnitzii | species | 单探针覆盖率90.62%,双探针覆盖率93.75% |
| 104 | G16F104-taxID1898203 | 乳酸菌科细菌 | Lachnospiraceae bacterium | species | 单探针覆盖率85.05%,双探针覆盖率94.66% |
| 105 | G16F105-taxID1265 | 黄面瘤胃球菌 | Ruminococcus flavefaciens | species | 单探针覆盖率88.89%,双探针覆盖率100% |
| 106 | G16F106-taxID163202 | 阑尾棒状杆菌 | Corynebacterium appendicis | species | 单探针覆盖率100% |
| 107 | G16F107-taxID796942 | 长口梭菌 | Stomatobaculum longum | species | 单探针覆盖率100% |
| 108 | G16F108-taxID437897 | 巨单形菌 | Megamonas funiformis | species | 单探针覆盖率100% |
| 109 | G16F109-taxID28131 | 中间普雷沃菌 | Prevotella intermedia | species | 单探针覆盖率93.65%,双探针覆盖率100% |
| 110 | G16F110-taxID28112 | 福赛坦纳菌 | Tannerella forsythia | species | 单探针覆盖率100% |
| 111 | G16F111-taxID1311 | 无乳链球菌 | Streptococcus agalactiae | species | 单探针覆盖率97.45%,双探针覆盖率100% |
| 112 | G16F112-taxID221279 | 候选竞争杆菌 ASV2 | Candidatus Competibacter ASV2 | genus | 单探针覆盖率100% |
| 113 | G16F113-taxID821 | 普通拟杆菌 | Bacteroides vulgatus | species | 单探针覆盖率90.90%,双探针覆盖率95.45% |
| 114 | G16F114-taxID549 | 聚集肠杆菌 | Pantoea agglomerans | species | 单探针覆盖率90.61%,双探针覆盖率98.12% |
| 115 | G16F115-taxID34038 | 水生拉恩氏菌 | Rahnella aquatilis | species | 单探针覆盖率95.55%,双探针覆盖率97.77% |
| 116 | G16F116-taxID103796 | 猕猴桃致病变种丁香假单胞菌 | Pseudomonas syringae pv. actinidiae | no rank | 单探针覆盖率98.48% |
| 117 | G16F117-taxID40323 | 嗜麦芽寡养单胞菌属 | Stenotrophomonas | genus | 单探针覆盖率90.71%,双探针覆盖率95.86% |
| 118 | G16F118-taxID48736 | 拉乌顿菌属 | Ralstonia | genus | 单探针覆盖率89.46%,双探针覆盖率96.28% |
| 119 | G16F119-taxID379 | 根瘤菌属 | Rhizobium | genus | 单探针覆盖率91.96%,双探针覆盖率98.42% |
| 120 | G16F120-taxID1873 | 小单孢菌属 | Micromonospora | genus | 单探针覆盖率93.10%,双探针覆盖率98.10% |
| 121 | G16F121-taxID41276 | 解磷短波单胞菌 | Brevundimonas vesicularis | species | 单探针覆盖率88.88%,双探针覆盖率100% |
| 122 | G16F122-taxID824 | 纤细弯曲菌 | Campylobacter gracilis | species | 单探针覆盖率88.88%,双探针覆盖率100% |
| 123 | G16F123-taxID294 | 荧光假单胞菌 | Pseudomonas fluorescens | species | 单探针覆盖率91.86%,双探针覆盖率96.27% |
| 124 | G16F124-taxID33035 | 球形布劳特氏菌 | Blautia coccoides | species | 单探针覆盖率100% |
| 125 | G16F125-taxID215200 | 拉氏消化链球菌 | Peptostreptococcus russellii | species | 单探针覆盖率100% |
| 126 | G16F126-taxID1509 | 产孢梭菌 | Clostridium sporogenes | species | 单探针覆盖率100% |
| 127 | G16F127-taxID1134687 | 密歇根克雷伯氏菌 | Klebsiella michiganensis | species | 单探针覆盖率88.52% |
| 128 | G16F128-taxID1309 | 变异链球菌 | Streptococcus.mutans | species | 单探针覆盖率96.93%,双探针覆盖率98.97% |
| 129 | G16F129-taxID354351 | 不解糖假苍白杆菌 | Pseudochrobactrum asaccharolyticum | species | 单探针覆盖率100% |
| 130 | G18F130-taxID45133 | 可可毛色二孢菌 | Lasiodiplodia theobromae | species | 单探针覆盖率100% |
下表列出了目前荧光显微镜中常见的4种荧光通道:DAPI、FITC、Texas Red和CY5的滤片波长范围,与其最匹配和兼容的荧光基团种类,同时注明了这些荧光基团的主要应用(如抗体标记、蛋白表达或FISH探针标记):FISH探针荧光标记的选择
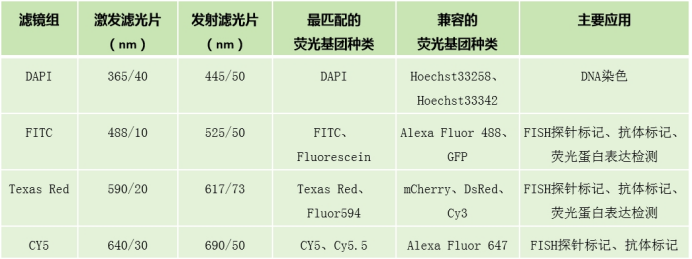
请注意,这些数值和建议的荧光基团是基于常见的荧光显微镜滤光片设置和荧光染料的特性在实际应用中,建议参考显微镜的用户手册、滤光片制造商的规格以及荧光染料供应商的信息,以确保最佳的荧光信号和成像效果。
FISH探针荧光标记策略推荐(请用户结合实际情况确认):
单种细菌16S FISH标记:
目标菌标记,优先选择标记Texas Red, 兼容CY3
阴性对照:推荐FITC标记 *微基生物FISH服务可免费提供
阳性对照(细菌通用探针):CY5 *微基生物FISH服务可免费提供
DAPI通道染DNA
双色FISH探针标记(同时检测两种菌,或使用两种不同荧光标记的探针检测同一种菌):
阳性对照(细菌通用探针):FITC *微基生物FISH服务可免费提供
目标菌1:推荐Texas Red,兼容CY3
目标菌2:推荐 CY5
DAPI通道染DNA
此种情况下,如需负对照,需在另一张片子中检测
产品特点
每款探针都经过严格设计和评测,确保高特异性和高覆盖率。
每款探针都附有详细的评测报告,包括特异性、覆盖率和应用建议。
单条FISH探针特异性较差的,通过设计双探针及多条探针提高特异性。
FISH 16S rRNA检测中使用双探针的好处:
增加特异性:由于16S rRNA基因在不同细菌中具有不同的保守区域和可变区域,使用双探针可以同时靶向这些区域,从而提高对目标菌种的特异性识别。
提高检测的灵敏度和准确性:双探针可以提供双重确认,减少因单一探针可能的非特异性结合导致的假阳性结果。
适用性广泛:双探针FISH技术可以应用于多种微生物的检测,包括细菌、古菌和真菌,使其成为一种多功能的微生物检测工具。
有助于微生物群落分析:在微生物群落结构分析中,双探针可以更精确地识别和区分群落中的不同成员,为微生物生态学研究提供重要信息。
欢迎访问我们的网站,了解更多关于16S/18S FISH探针的详细信息,以及如何将这些强大的工具应用到您的研究中。微基生物科技,与您携手共创科研新篇章。

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用




