菌株比较基因组分析是通过对不同菌株的全基因组进行测序和比较,揭示菌株之间的遗传差异、进化关系及功能特性关联的一种生物技术手段。微基生物依托先进测序技术与生物信息学分析方法,推出菌株比较基因组检测分析服务,提供样本处理、高通量测序、到深度数据分析的全流程一站式解决方案。
服务流程
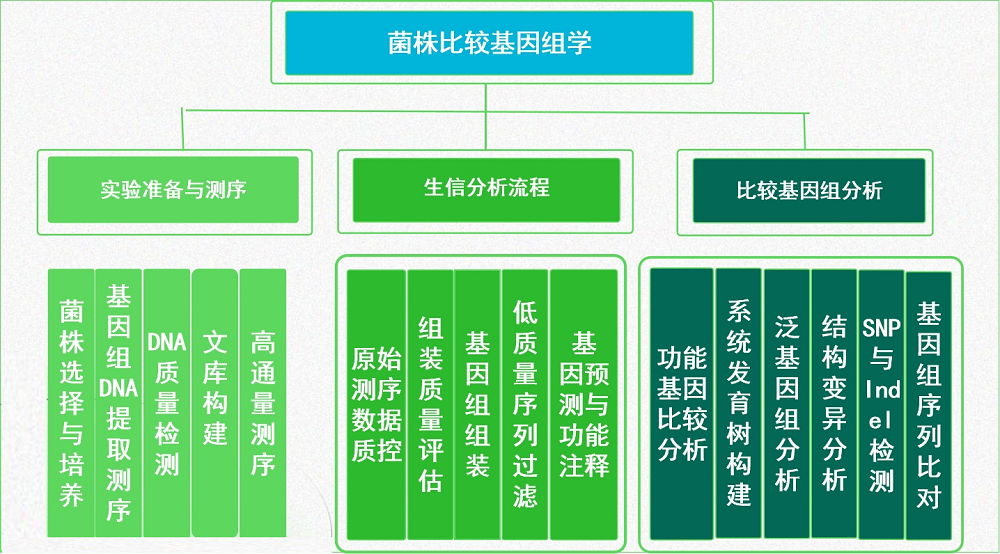
微基生物提供比较基因组分析:基因组序列比对、系统发育树构建、泛基因组分析、结构变异分析、功能基因(如抗生素抗性基因、代谢相关基因)检测等,全方位解析菌株遗传特征。
适用场景
1.新菌株鉴定:未知菌株与已知菌株基因组比对,快速确定新菌株分类地位和独特遗传特征,为新菌株的资源保护、开发利用(如新型益生菌、工业菌株筛选)提供核心依据。
2.菌株进化研究:无论是探究同一物种不同生态型菌株的进化路径,还是研究菌株在不同环境压力下的适应性进化,通过系统发育分析、遗传变异解析等手段,清晰还原菌株进化历史,揭示遗传变异与环境适应的关联机制,为微生物进化生物学研究提供有力支撑。
3.功能基因挖掘:可定向挖掘与抗生素抗性、特殊代谢产物(如有机酸、酶制剂)、污染物降解等相关的功能基因,为后续研究奠定基础。
结果可视化化图表展示
一、不同生态位植物乳杆菌(Lactiplantibacillus plantarum)比较基因组分析
1.研究背景
植物乳杆菌(Lactiplantibacillus plantarum)广泛存在于植物、乳制品、肉制品和胃肠道等环境中,具有益生功效。该研究旨在通过比较基因组分析,揭示不同生态位来源的植物乳杆菌的基因组特征差异,探索其生态适应性机制。
2.样本来源
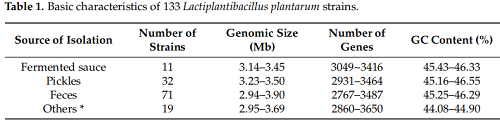
分离菌株:采集324份样品(126份泡菜、32份发酵酱、166份人类粪便),分离获得114株植物乳杆菌,结合NCBI数据库19株参考菌株,共133株进行比较分析。
参考菌株:从NCBI数据库选取19株全基因组完整的植物乳杆菌参考菌株,来源涵盖酸菜、婴儿粪便、酿造环境、唾液、乳制品等,总计133株菌株进行分析。
3.分析内容
全基因组测序、基因组组装与注释、比较基因组分析、基因型与表型关联分析,重点探究核心基因与独特基因分布、系统发育关系、碳水化合物活性酶(CAZy)家族差异等。
4.关键结果与可视化展示
(1)基因分布特征
133株植物乳杆菌基因组大小为2.94-3.90Mb,基因数量2767-3650个,GC含量44.08%-46.55%。其中,粪便源菌株的基因组大小与基因数量变异范围更宽,提示其遗传多样性更高;参考菌株GC含量(44.08%-44.90%)整体低于分离菌株,可能与长期实验室培养驯化相关。
(2)核心基因与独特基因分布
从133株菌株中鉴定出1620个核心基因,这些基因是植物乳杆菌维持基本生命活动(如DNA复制、能量代谢、氨基酸合成)的关键,且核心基因数量高于其他乳酸菌(如乳球菌),解释了其对不同生态位的强适应性。同时,不同分离源菌株拥有丰富的独特基因:粪便源菌株平均独特基因数量最多,发酵酱源菌株最少,这些独特基因与菌株适应特定生态位(如肠道碳水化合物代谢、食品基质降解)密切相关。
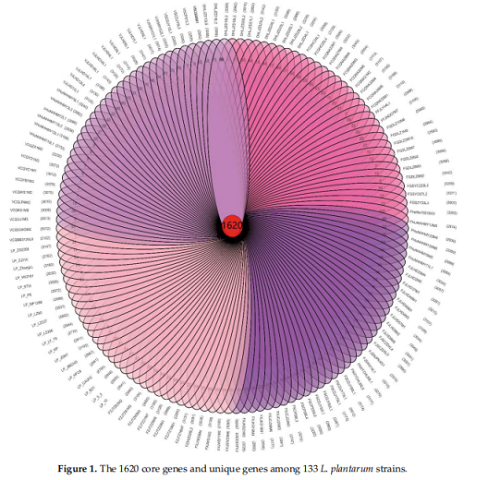
133 株植物乳杆菌核心基因与独特基因分布
(3)系统发育关系
基于1620个核心基因序列构建的系统发育树显示:133株菌株分为A、B两大簇,A簇仅含6株菌株(粪便源与泡菜源混合),因菌株数量少未呈现明显生态位聚类;B簇含127株菌株,进一步分为B-1至B-6亚簇,其中泡菜源与发酵酱源菌株聚类明显(如8株发酵酱源菌株集中在B-1亚簇,8株泡菜源菌株集中在B-6亚簇);粪便源菌株在B簇各亚簇中均有分布,无特定聚类模式,推测人类肠道中的植物乳杆菌可能来源于多样化的食物(如泡菜、发酵酱),经饮食摄入后适应肠道环境;参考菌株根据其来源分散在不同分支,如乳制品源参考菌株与食品源分离菌株聚类,婴儿粪便源参考菌株与成人粪便源分离菌株聚类,进一步验证生态位对菌株进化的影响。
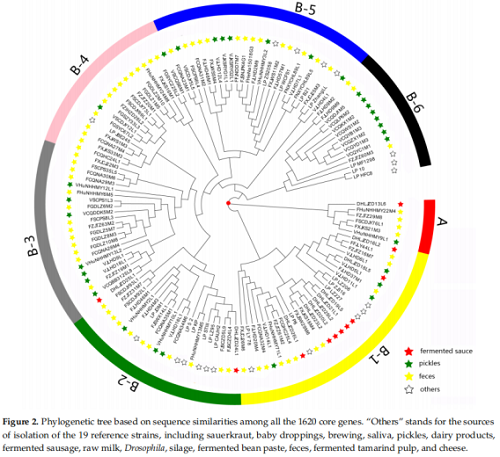
基于 1620 个核心基因的植物乳杆菌系统发育树
(4)平均核苷酸一致性(ANI)分析
ANI是判断菌株是否为同一物种的核心指标(通常阈值96%),研究中泡菜源菌株间ANI值普遍>99%(如重庆泡菜源8株菌株ANI值99.23%),发酵酱源菌株间 ANI值>99%(如黑龙江发酵酱源菌株),表明同一食品来源菌株遗传相似度极高;泡菜源与发酵酱源菌株间ANI值98.85%-99.02%,低于同来源菌株但高于种水平阈值,提示两者虽为同一物种,但因长期适应不同食品基质(植物纤维vs蛋白质)产生遗传分化;A簇6株菌株与B簇127株菌株间最大ANI值仅95.23%,接近但低于96%阈值,推测A簇菌株可能为植物乳杆菌属内的独立亚种,需进一步实验验证。

133株植物乳杆菌ANI热图
(5)碳水化合物活性酶(CAZy)与碳水化合物利用关联
对114株分离菌株的碳水化合物活性酶(CAZy)进行预测,发现不同分离源菌株的CAZy家族分布存在明显差异(“F”粪便、“V”泡菜、“D”发酵酱)。例如,粪便来源菌株可能含有更多适应肠道碳水化合物代谢的CAZy酶,发酵食品来源菌株可能富集与食品基质降解相关的酶类,为植物乳杆菌的功能应用场景选择提供了直接依据。
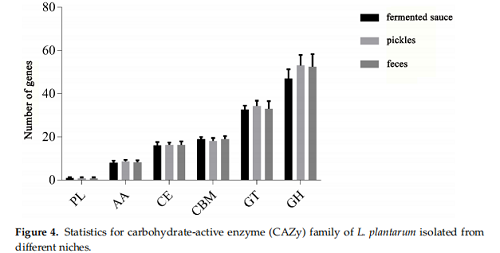
不同来源植物乳杆菌CAZy家族基因数量统计
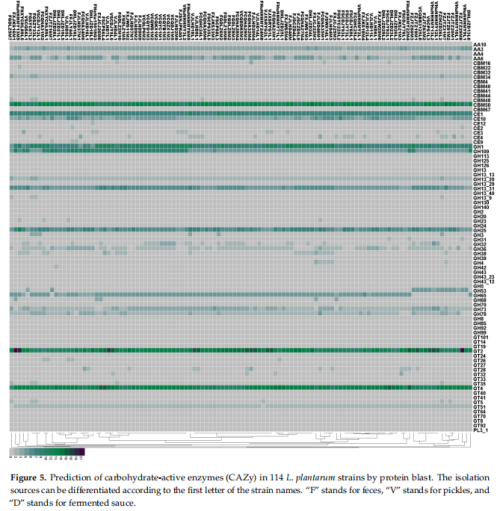
114 株植物乳杆菌碳水化合物利用热图
二、南极企鹅粪便来源琼脂降解菌(Flavobacterium faecale WV33ᵀ)比较基因组分析
1.研究背景
琼脂糖降解菌在食品工业、生物医药、环境治理等领域具有重要的应用价值,其产生的琼脂酶可用于琼脂糖的降解与转化。Agarolytic Flavobacterium faecale WV33T是一株具有琼脂糖降解功能的菌株,对其进行比较基因组分析,有助于深入了解其耐药性及琼脂糖降解机制,为该菌株的开发利用提供科学依据。
2.样本来源
目标菌株:F.faecale WV33ᵀ,分离自南极企鹅粪便,严格好氧、非运动性杆状菌,最适生长温度16℃,产橙色色素(玉米黄质);
参考菌株:NCBI数据库选取26株黄杆菌属(Flavobacterium)模式菌株,来源涵盖淡水、海水、土壤、鱼体等,总计27株菌株进行比较分析。
3.核心分析内容
全基因组测序与组装、基因组注释、系统发育与ANI分析、功能基因验证。
4.关键结果与可视化展示:
(1)基因组基本特征
F.faecale WV33ᵀ基因组为单一环状染色体,大小4,621,116 bp,GC含量35.2%,含3984个编码DNA序列(CDS)、18个rRNA 基因(5S/16S/23S)及67个tRNA基因,基因密度885个/Mb。COG 功能分类显示,“碳水化合物运输与代谢”(183个CDS,7.1%)、“氨基酸运输与代谢”(170个 CDS,6.6%)、“细胞壁/膜/包膜生物发生”(260个CDS,10.1%)相关基因占比高,与菌株降解多糖、适应南极极端低温环境的功能需求相契合。
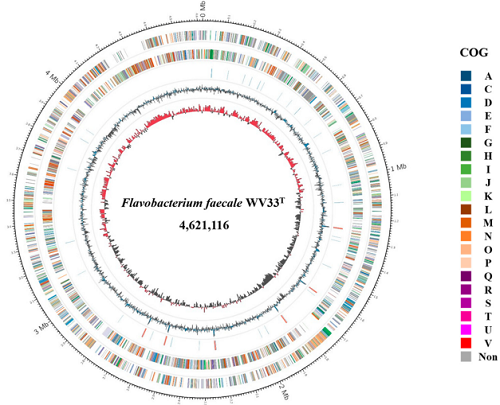
F. faecale WV33ᵀ基因组环状图
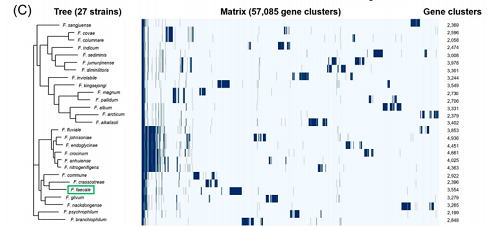
(2)菌株分类与亲缘关系
ANI分析显示,F. faecale WV33ᵀ与26株黄杆菌属模式菌株的ANI值为 0.66-0.91(ANIblastall 算法),均低于96%的种水平阈值,明确其为黄杆菌属中的独立新物种;系统发育树进一步表明,WV33ᵀ与南极企鹅粪便来源的Flavobacterium kingsejongi亲缘关系最近(分支距离最短),推测两者在极端低温环境(南极)中共同进化,形成相似的遗传适应特征(如低温适应基因)。
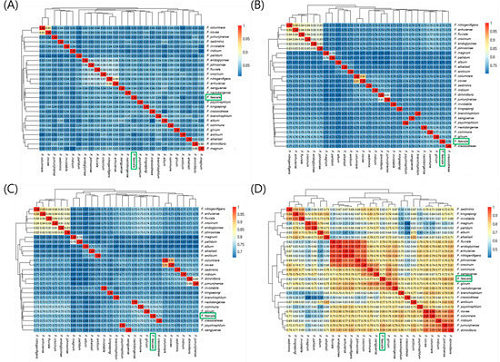
27 株黄杆菌属菌株 ANI 热图(ANIblastall 算法)

基于 90 个单拷贝基因的黄杆菌属系统发育树
(3)耐药性基因分析
通过基因组注释与ResFinder数据库比对,未发现F.faecale WV33ᵀ携带常见高风险耐药基因(如β-内酰胺类、氨基糖苷类、四环素类耐药基因),仅含1个低风险多重耐药外排泵基因(emrB),且该基因在黄杆菌属中普遍存在,无特殊耐药性。这一结果表明,WV33ᵀ在食品、生物医药领域应用时,不存在耐药基因水平转移的安全隐患,具备良好的应用安全性。
(4)琼脂糖降解机制解析
在WV33ᵀ基因组中鉴定出7个putative琼脂酶基因(agar.3、agar.162、agar.2965、agar.3018、agar.3061、agar.3068、agar.3154),均含完整开放阅读框与GH16家族功能结构域(琼脂酶核心结构域)。RT-qPCR分析显示,在以琼脂为唯一碳源的培养基中5个琼脂酶基因(agar.3、agar.2965、agar.3018、agar.3061、agar.3068)的表达量显著上调,其中agar.3061表达量最高(是葡萄糖组的14倍);基因敲除实验进一步证实,agar.3018与 agar.3061敲除后,菌株琼脂糖降解活性下降70%-80%,而互补菌株可恢复活性,表明这两个基因为 WV33ᵀ琼脂糖降解的关键基因,为后续基因工程改造(如提高琼脂酶产量)提供明确靶点。
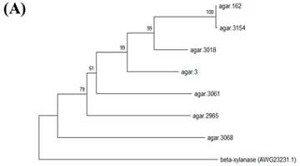
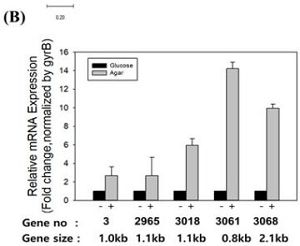
F.faecale WV33ᵀ琼脂酶基因表达与功能验证
(5)泛基因组分析
对27株黄杆菌属菌株进行泛基因组分析,结果显示:核心基因数量仅294个(占总基因簇的0.5%),随菌株数量增加核心基因数量快速下降并趋于稳定,表明黄杆菌属菌株间核心功能基因较少,遗传差异较大;泛基因组基因数量随菌株数量增加持续上升,无平台期,表明黄杆菌属为“开放泛基因组”,菌株间存在广泛的基因获得与丢失,可能通过水平基因转移、噬菌体整合等方式获取新基因,以适应不同生态环境;WV33ᵀ含2369个特有基因,其中包括4个琼脂酶基因与3个次级代谢产物基因簇,进一步体现其独特的功能潜力。
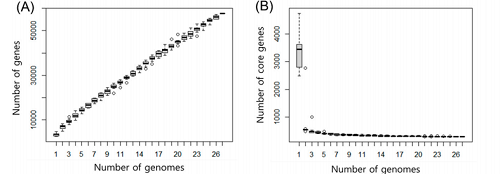
27株黄杆菌属菌株泛基因组与核心基因组分析
无论是探索微生物进化奥秘、挖掘功能基因资源,还是推动菌株产业化应用,微基生物菌株比较基因组检测分析服务都将是您的可靠伙伴。我们期待与您携手,以先进的基因组技术赋能微生物研究与产业创新,共同开启微生物资源开发的新未来!
参考文献:
1.Mao B Y, Yin R M, Li X S, et al. Comparative Genomic Analysis of Lactiplantibacillus plantarum Isolated from Different Niches[J]. Genes, 2021, 12(2): 241.
2.Lee J H, Lee S R, Han S, et al. Comparative Genomic Analysis of Agarolytic Flavobacterium faecale WV33T[J]. International Journal of Molecular Sciences, 2022, 23(18): 10884.

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用


-1.jpg)
-2.jpg)
-3原始.jpg)

