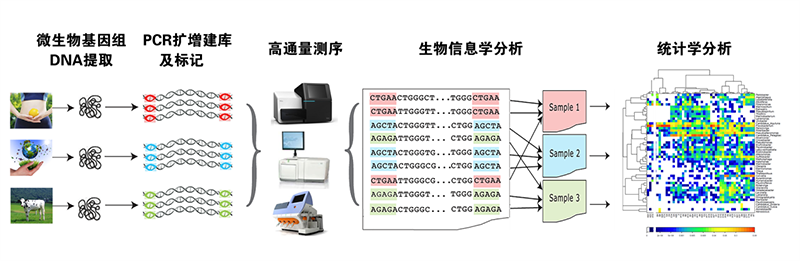
微基生物提供根系微生物多样性分析的整体科研服务:实验规划->样本采集保存->分子实验->生信统计分析->论文协助
微基生物采用高通量测序、PCR-DGGE、实时荧光定量PCR等方法,对样本中的DNA进行序列测定,并通过生信统计分析,对大量数据进行处理,揭示肠道中微生物的种类以及它们之间的相对丰度和进化关系,探讨微生物多样性,研究根系微生物与环境间的相关关系。
技术路线:
高通量分析流程
PCR-DGGE分析流程
检测平台:
微基生物拥有Illumina MiSeq、Ion PGM、Roche 454高通量测序分析,PacBio第三代高通量测序分析,PCR-DGGE变性梯度凝胶分析,实时荧光定量PCR(Real-time qPCR),克隆文库等检测平台。
样品采集:
微基生物为客户提供样品采集的配套工具,如采集盒、保存液、取样勺和保存管等。
送样要求:
样品原样
(1)样品类型:根系微生物,新鲜取样,冻存于-80℃
(2)样品需求:≥2g
(3)样品保存期间切忌反复冻融,送样时请使用冰袋或干冰运输
DNA类型
(1) 样品类型: DNA
(2) 样品需求量:≥300ng
(3) 样品浓度: ≥10ng/μL
(4) 样品纯度:OD260/280=1.8-2.0并确保DNA无降解
(5) 样品保存期间切忌反复冻融,送样时请使用冰袋或干冰运输
(6) 对于本种类型的样品,我们在检测完样品的质量后,进行PCR扩增等后续试验
生物信息与统计学服务:
生信分析项目
更多微生态方向研究和生物信息方面服务,请详询:400-660-9270
标题:运用PCR-DGGE和焦磷酸测序分析(Roche 454)对红树林湿地和根围古菌的多样性分析
研究领域:根系微生物
分析物种:古菌
研究区域:16S rRNA V4 and V5 regions
研究方法:PCR-DGGE和Roche 454
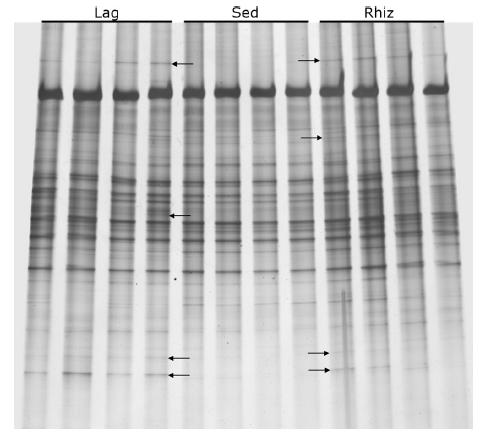
主要结果:
- 实验DGGE结果
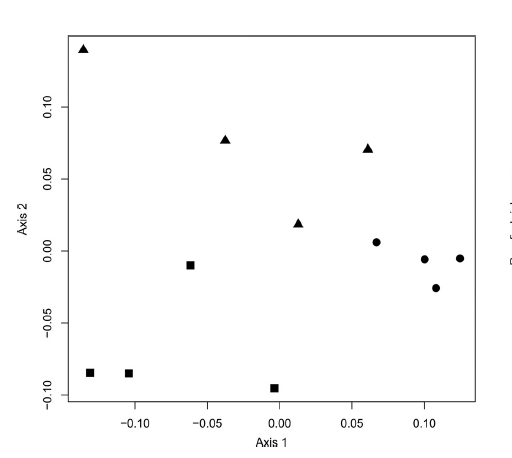
 2.两个PCO轴和展示了所有数据集58%的变种,(根围上、中、下古菌菌群的展示)
2.两个PCO轴和展示了所有数据集58%的变种,(根围上、中、下古菌菌群的展示)
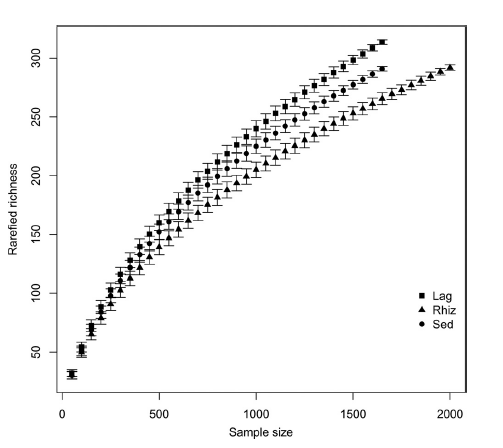
 3.采用重测样对16S rRNA的样本宽度和样本测序丰富度曲线
3.采用重测样对16S rRNA的样本宽度和样本测序丰富度曲线
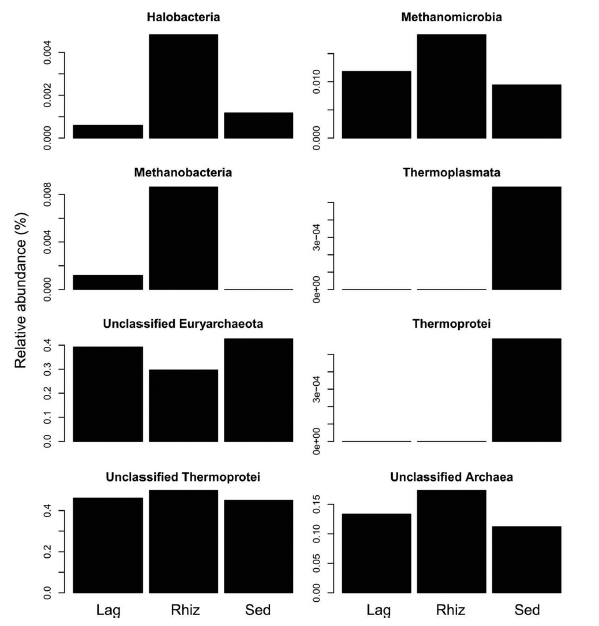
4.运用RDP的分类器算法统计出古菌16S rRNA在三个不同地方的差异
5.根际微生物中不同地方占主导地位OUT的相关关系
6.被检索到的古菌序列的具体分类
原文链接:http://www.ncbi.nlm.nih.gov/pubmed/22660713
参考文献:
Pires, A. C., D. F. Cleary, A. Almeida, A. Cunha, S. Dealtry, L. C. Mendonca-Hagler, K. Smalla and N. C. Gomes (2012). “Denaturing gradient gel electrophoresis and barcoded pyrosequencing reveal unprecedented archaeal diversity in mangrove sediment and rhizosphere samples.” Appl Environ Microbiol 78(16): 5520-5528.

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用