1 检测对象
特定功能基因指的是具有某种特定作用的酶的编码基因,通过对该酶的基因进行qPCR定量,来确定该基因在样本中的拷贝数。
目前研究较多的主要是氮循环过程中每个反应的酶,碳循环,硫相关,砷相关的一些酶的拷贝数信息。
氮循环相关
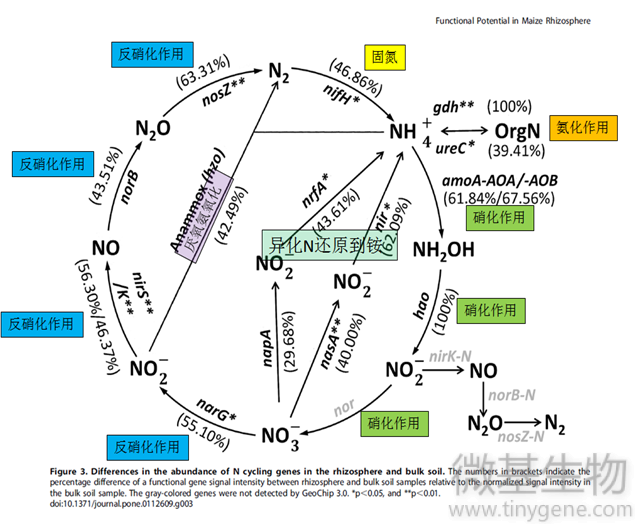
氮是生命体核酸与蛋白质必不可少的组成元素。氮素的生物地球化学循环(氮循环)对生命的存在和持续有关键作用,本质上是微生物驱动的氮素转化、利用及循环的过程。氮循环就是指N2、无机氮化合物、有机氮化合物在自然界中相互转化过程的总和,包括氨化作用、硝化作用、反硝化作用、固氮作用等等。
氮循环及相关基因如下图和下表:
| 参与途径 | 基因名称 | |||
| 固氮 | N2—NH4+ | nifH | Nitrogenase | 固氮酶 |
| 氨化作用 | OrgN—NH4+ | ureC | Glutamate dehydrogenase | 谷氨酸脱氢酶 |
| 硝化作用 | NH4+—NO2– | amoA | ammonia monooxygenase | 氨单加氧酶 |
| NO2–—NO3– | nxrA | nitrite oxidoreductase | 亚硝酸盐氧化还原酶 | |
| 反硝化作用 | NO3–—NO2– | narG | nitrate reductase | 硝酸盐还原酶 |
| NO2–—NO | nirS/nirK | nitrite reductase | 亚硝酸盐还原酶 | |
| NO—NO2 | norB | nitric oxide reductase | 氧化还原酶 | |
| NO2—N2 | nosZ | nitrous oxide reductase | 氧化亚氮还原酶 | |
| 厌氧氨氧化 | NO2–—N2 | hzo | Hydrazine oxidase | 联氨氧化酶 |
| 异化N还原到铵 | NO3–—NO2– | napA | nitrate reductase | 硝酸盐还原酶 |
| NO2–—NH4+ | nrfA |
c-type cytochrome nitrite reductase |
细胞色素c亚硝酸盐还原酶 | |
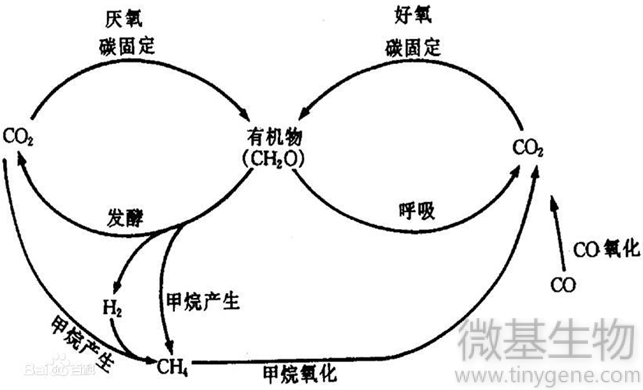
碳循环相关
碳循环主要是碳素的固定和转化过程,主要包括碳固定、甲烷产生、甲烷氧化。
自养微生物具有极强的环境适应性和固碳潜力。目前发现的5条主要生物固碳途径中,卡尔文循环是自养生物固定CO2的主要途径,其中核酮糖-1,5-二磷酸羧化酶/加氧酶(RubisCO)是卡尔文循环中的关键酶,因此RubisCO及其编码基因被许多学者用于不同生态环境中固碳微生物群落结构和多样性的研究。
产甲烷菌是重要的环境微生物,在自然界的碳素循环中起重要作用。产甲烷菌是一类能够将无机或有机化合物厌氧发酵转化成甲烷和二氧化碳的古细菌。
甲烷氧化菌是甲烷进入大气的重要屏障,利用它降低人为源向大气排放的甲烷量,对于缓解全球温室效应具有潜在价值.
碳循环过程和相关基因如图和下表:
| 作用 | 基因名称 | |||
| 碳相关 | 固碳 | cbbL/cbbM |
Ribulose-l,5-bisphosphate carboxylase/oxygenase |
核酮糖-1.5-二磷酸羧化酶/加氧酶 |
| 产生甲烷 | mcrA | methyl coenzyme M reduc-tase | 甲基辅酶 M 还原酶 | |
| 甲烷氧化 | pmoA | particulate methane monooxygenase | 甲烷氧化单加氧酶 | |
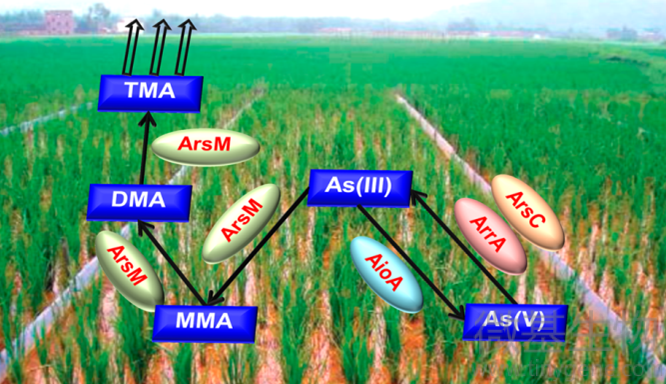
硫相关和砷相关的检测基因见下表:
| 作用 | 基因名称 | |||
| 硫相关 | 硫酸盐还原 | dsrA | dissimilatory sulfite reductase | 异化型亚硫酸盐还原酶 |
| dsrB | dissimilatory sulfite reductase | 异化型亚硫酸盐还原酶 | ||
| 硫氧化 | soxB | Sulfur oxidation | 硫氧化酶 | |
| 砷相关 | 砷氧化 | aioA | arsenite (As(III)) oxidase genes | 亚砷酸盐氧化酶基因 |
| 砷呼吸还原 | arrA | respiratory arsenate (As(V)) reductase genes | 呼吸性砷酸盐还原酶基因 | |
| 砷解毒还原 | arsC | As(V)reductase genes | 砷还原酶基因 | |
| 砷甲基化 | arsM | As(III) methyltransferase genes | 砷甲基转移酶基因 | |
2 qPCR的原理
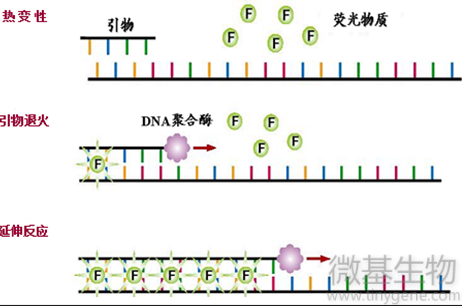
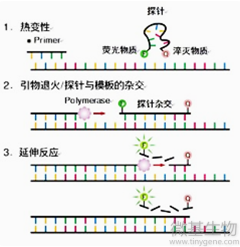
实时荧光定量PCR技术(Real-timeQuantitativePCR,qPCR)是指在PCR反应体系中加入可与DNA产物特异性结合的荧光基团,利用荧光信号积累实时监测整个PCR进程,最终通过相对定量(与内参基因对比)或绝对定量(通过标准曲线对未知模板进行定量)的方法确定各个样本的本底表达量。
3 3种荧光试剂的工作原理及区别
SYBRGreenⅠ法:
嵌入到双链DNA分子后在PCR反应体系中,加入过量SYBR荧光染料,SYBR荧光染料特异性地掺入DNA双链后构象发生变化,能够吸收497nm的激发光并发出520nm的荧光;而不掺入DNA双链中的染料分子不会发射任何荧光信号,从而保证荧光信号的增加与PCR产物的增加完全同步。
TaqMan探针法:
扩增时加入一个特异性的寡核苷酸荧光探针,两端分别标记一个报告荧光基团和一个淬灭荧光基团。探针完整时,报告基团发射的荧光信号被淬灭基团吸收;PCR扩增时,Taq酶的5′-3’外切酶活性将探针酶切降解,使报告荧光基团和淬灭荧光基团分离,从而荧光监测系统可接收到荧光信号,即每扩增一条DNA链,就有一个荧光分子形成,实现了荧光信号的累积与PCR产物形成完全同步
SYBRGREEN法和TaqMan探针法的区别
| 方法 | 优点 | 缺点 |
|
SYBR GREEN法 |
高灵敏度,操作简单,不影响酶促反应,价格便宜 |
1与探针法相比,对引物特异性要求较高,需进行熔解曲线分析 2灵敏度相对较低。 |
|
TaqMan 探针法 |
1具有高度特异性 2更高的灵敏度 3可同时检测几种不同的荧光信号的产物 |
1探针价格较高 2需要不同引物的扩增效率一致,对引物的设计及扩增条件要求比较高 |
4 qPCR的实验方法
| 实验方法 | 原理 | 技术应用 | 相关应用 |
| 绝对定量(AbsoluteQuantification,AQ) | 是测定目的基因在样本中的分子数目,通过构建标准曲线对未知模板进行分子数目的定量,即通常所说的拷贝数。 |
特定微生物的拷贝数检测 特定功能基因的拷贝数检测 特定抗性基因的拷贝数检测 |
1临床疾病诊断 2动物疾病检测 3食品安全 4科学研究 5应用行业:各级各类医疗机构、大学及研究所、CDC、检验检疫局、兽医站、食品企业及乳品厂等。 |
| 相对定量(RelativeQuantification,RQ) | 测定目的基因在样本中的含量的相对比例,而不需要知道它们在每个样本中的绝对拷贝数,一般是通过CT值之差来计算。 |
样本中某种特定微生物拷贝数占全部微生物拷贝数的比例 某种功能基因的微生物拷贝数占全部微生物拷贝数的比例 某种抗性基因拷贝数占全部微生物拷贝数的比例 |
|
相对定量和绝对定量的数据计算方法
标准质粒拷贝数计算:
每微升拷贝数(copies/μl)=[质粒浓度(ng/ul)×10-9×6.02×1023]/克隆产物相对分子质量
原始样本目标基因拷贝数copies/μLDNA=拷贝数*稀释倍数
原始样本目标基因拷贝数copies/g样本=(原始样本目标基因拷贝数copies/μLDNA*基因组DNA体积)/样本抽提重量,(如果客户样本为DNA样本,则不需要计算copies/g样本)
相对定量的计算:
步骤1:内参基因均一化样本差异:Ct目的基因–Ct内参基因=△Ct
步骤2:其他样本和对照样本比较:△Ct处理样本–△Ct对照样本=△△Ct
步骤3:使用公式计算:倍数变化=2-△△Ct
5 qPCR的实验流程
6 qPCR检测送样要求
| 样本类型 | 样品量/例 | 备注 | |
| 环境 | 土壤/沉积物/淤泥 | 1g | |
| 湖水/海水/河水 | 1L,用滤膜或离心富集 | 过 0.22μm 的滤膜,或者 12000rpm 离心,进行富集。 | |
| 污水 | 20ml | 若样本清亮则可适当地多取。 | |
| 泥水混合样 | 2ml | ||
| 空气 | 根据实验需要,用无菌滤膜过滤空气。 | ||
| 发酵物 | 固体 2g,液体 20ml |
| 样本类型 | 样品量/例 | 备注 | |
| 人体 | 粪便 | ≥3g | |
| 皮肤 | 5 个采集拭子 | 采集 5cm*5cm 面积,反复刮取 20 次。 | |
| 生殖道 | 5 个采集拭子 | 采集阴道口内 4cm 处分泌物, 每个拭子转 3 圈。 | |
| 牙菌斑/舌苔 | 5 个采集拭子 | 在采集的部位,刮取 10 次左右。 | |
| 唾液 | 10ml | ||
| 痰液 | 10ml 或 2-3 口痰液 | ||
| 鼻腔 | 5 个采集拭子 | 采集鼻腔内粘膜上的分泌物,转 3 圈。 | |
| 肺部灌洗液 | 30ml 富集液 | 对灌洗液进行离心富集。 | |
| 肠道/胃组织 |
5mm*5mm*3mm 3 块 |
尽量多一取些。 | |
| 血液 | 3ml 全血 | 用 EDTA 抗凝管,颠倒 8-10 次。 不建议用肝素。 | |
| 尿液 | 30mL 尿液 | 取中段尿为宜。 | |
| 母乳 | 5mL | ||
| 动物 | 粪便 | ≥3g | 最少 0.05g。 |
|
盲肠/结肠/胃 组织 |
≥3g | 至少 1g,如果实验条件允许,尽可能多的收集样本 | |
| 肠道内容物 | ≥3g | 最少 0.05g。建议客户自己取内容物,可以用 PBS 冲洗。 |
提醒
1 因为qPCR绝对定量最终得到的是单位重量,或是单位体积目标基因的拷贝数,所以如果客户送DNA,建议客户记录抽提DNA时样本的使用重量或是体积,方便后继对数据进行转化。
2 送样时请尽量使用冰盒(泡沫盒+干冰),以保证运输过程中的低温条件(干冰的挥发消耗量约为 3-4 公斤/天)。在打包时放入足量的干冰, 将样本埋入干冰中, 为了保持冰盒中的低温环境,建议采用加厚的泡沫盒
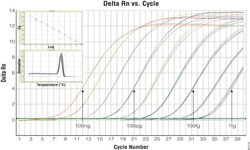
7 qPCR结果
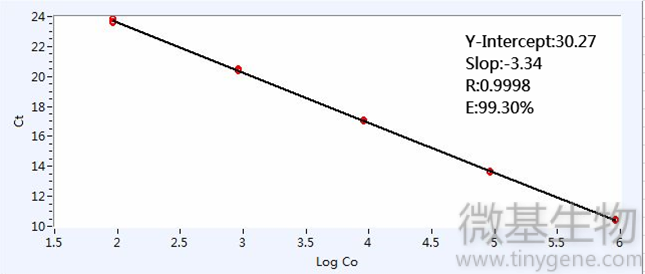
标准曲线
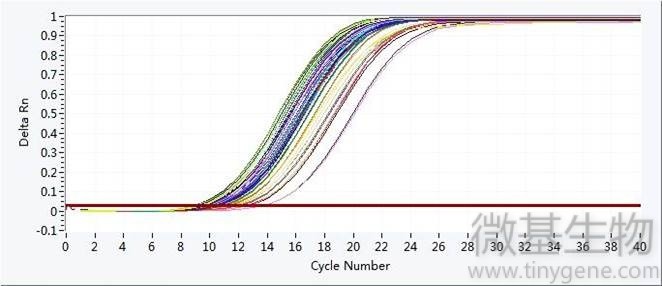
扩增曲线
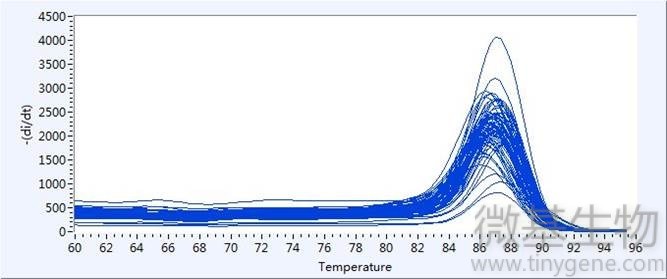
熔解曲线
定量结果
| 微基编号 | 拷贝数 | 样本稀释倍数 | 抽提样本重量(g) | 基因组DNA体积(μL) | 原始样本目标基因拷贝数copies/μL DNA | 原始样本目标基因平均拷贝数copies/μL DNA | 原始样本目标基因拷贝数copies/g样本 | 原始样本目标基因平均拷贝数copies/g样本 |
| 1 | 1.08E+06 | 200 | 0.237 | 50 | 2.17E+08 | 2.26E+08 | 4.58E+10 | 4.76E+10 |
| 1 | 1.23E+06 | 200 | 0.237 | 50 | 2.47E+08 | 5.20E+10 | ||
| 1 | 1.07E+06 | 200 | 0.237 | 50 | 2.14E+08 | 4.51E+10 | ||
| 2 | 7.28E+05 | 200 | 0.24 | 50 | 1.46E+08 | 1.49E+08 | 3.03E+10 | 3.09E+10 |
| 2 | 7.55E+05 | 200 | 0.24 | 50 | 1.51E+08 | 3.15E+10 | ||
| 2 | 7.44E+05 | 200 | 0.24 | 50 | 1.49E+08 | 3.10E+10 | ||
| 3 | 1.24E+06 | 200 | 0.25 | 50 | 2.48E+08 | 2.59E+08 | 4.96E+10 | 5.19E+10 |
| 3 | 1.35E+06 | 200 | 0.25 | 50 | 2.69E+08 | 5.39E+10 | ||
| 3 | 1.30E+06 | 200 | 0.25 | 50 | 2.61E+08 | 5.21E+10 | ||
| 4 | 1.61E+06 | 200 | 0.234 | 50 | 3.21E+08 | 3.22E+08 | 6.87E+10 | 6.87E+10 |
| 4 | 1.60E+06 | 200 | 0.234 | 50 | 3.19E+08 | 6.82E+10 | ||
| 4 | 1.62E+06 | 200 | 0.234 | 50 | 3.24E+08 | 6.93E+10 | ||
| 5 | 1.27E+05 | 200 | 0.175 | 50 | 2.54E+07 | 2.66E+07 | 7.25E+09 | 7.59E+09 |
| 5 | 1.33E+05 | 200 | 0.175 | 50 | 2.66E+07 | 7.61E+09 | ||
| 5 | 1.39E+05 | 200 | 0.175 | 50 | 2.77E+07 | 7.91E+09 |
8 术语解释
Ct值(Cyclethreshold,循环阈值):每个反应管内的荧光信号到达设定阈值时所经历的循环数。
荧光阈值(threshold)的设定:PCR反应的前15个循环的荧光信号作为荧光本底信号,荧光阈值的缺省(默认)设置是3-15个循环的荧光信号的标准偏差的10倍,即:threshold=10*SDcycle3-15
基线(baseline):在PCR扩增反应的最初数个循环里,荧光信号变化不大,接近一条直线,即称为基线。
熔解曲线Tm(TmOfMeltingCurve):SYBRGreen染料发实验结束后需对qPCR产物加热,随着温度的升高,双链接扩增产物逐渐解链,导致荧光强度下降,到达某一温度时,会导致大量的产物解链,荧光急剧下降,将此温度称为Tm值。不同PCR产物Tm值不同,从而可对PCR的特异性进行鉴定。
9 参考文献
1 Dan Chen et.al. Microbial community and metabolism activity in a bioelectrochemical denitrification system under long-term presence of p-nitrophenol.2018.Bioresource Technology.(narG nirK nirS napA)
2 Lei Wang et.al. Responses of bacterial and archaeal communities to nitrate stimulation after oil pollution in mangrove sediment revealed by Illumina sequencing.2016.Marine Pollution Bulletin.(nirK nirS)
3 Hailu Wu et.al . Effects of root exudates on denitrifier gene abundance, community structure and activity in a micro-polluted constructed wetland. 2017.ScienceoftheTotalEnvironment. (nirK nirS)
4 Suresh Kumar Dubeyet.al. Methane production potential and methanogenic archaeal community structure in tropical irrigated Indian paddy soils. 2014.Biol Fertil Soils.(mcrA)
5 Juanli Yun et.al. Diversity, abundance and vertical distribution of methane-oxidizing bacteria (methanotrophs) in the sediments of the Xianghai wetland, Songnen Plain, northeast China .2011.JSoilsSediments.(pmoA)
6 Hongxia Du et.al.Mercury-methylatinggenes dsrB and hgcA insoils/sedim entsofthe Three Gorges Reservoir.2016.Environment Scientifi cPollution Research.(dsrB)
7 Si-Yu Zhang et.al. Si-Yu Zhang Diversity and Abundance of Arsenic Biotransformation Genes in Paddy Soils from Southern China.2015. Enveronmental Science Technology.(砷相关基因)

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用