1 检测对象
关于特定种类的微生物,客户可告知物种信息及引物信息,微基生物针对目标微生物进行qPCR定量分析;也可告知感兴趣的物种名称,微基生物来查询相关引物信息;如老师感兴趣的物种信息尚未见文献报道,微基生物可以开发程序,自行研发寻找目标菌株的特异基因并设计目标菌株的特异引物,验证引物的特异性,进行后继实验。
微基生物已检测过的物种信息,见表格(仅列出了常见的部分微生物种类),其他的微生物也可以检测。
| 级别 | 中文名称 | 英文名称 |
| 界 | 细菌 | Bacteria |
| 界 | 真菌 | Fungi |
| 界 | 古菌 | Archaea |
| 门 | 放线菌门 | Actinobacteria |
| 门 | 拟杆菌门 | Bacteroidetes |
| 门 | 厚壁菌门 | Firmicutes |
| 纲 | alpha变形菌纲 | Alpha-proteobacteria |
| 纲 | gamma变形菌纲 | Gamma-proteobacteria |
| 科 | 肠杆菌科 | Enterobacteriaceae |
| 科 | 瘤胃球菌科 | Ruminococcaceae |
| 属 | 双歧杆菌属 | Bifidobacterium |
| 属 | 大肠杆菌属 | Escherichia |
| 属 | 乳酸杆菌属 | Lactobacillus |
| 属 | 梭菌属 | Clostridium |
| 属 | 肠球菌属 | Enterococcus |
| 属 | 拟杆菌属 | Bacteroides |
| 属 | 奇异菌属 | Atopobium cluster |
| 属 | 链球菌属 | Streptococcus |
| 属 | 瘤胃球菌属 | Ruminococcus |
| 属 | 粪杆菌属 | Faecalibacterium |
| 属 | 布劳特菌属 | Blautia |
| 属 | 艰难梭菌属 | Clostridium difficile |
| 属 | 脱硫弧菌属 | Desulfovibrio |
| 属 | 另支菌属 | Alistipes |
| 属 | 链球菌属 | Streptococcus |
| 属 | 萨特氏菌属 | Sutterella |
| 属 | 丙酸杆菌属 | Propionibacterium |
| 属 | 假单胞菌属 | Pseudomonas |
| 属 | 噬酸菌属 | Acidovorax |
| 属 | 不动杆菌属 | Acinetobacter |
| 属 | 鞘氨醇单胞菌属 | Sphingomonas |
| 属 | 分枝杆菌属 | Mycobacterium |
| 属 | 普氏菌属 | Prevotella |
| 种 | 溶纤维丁酸弧菌 | Butyrivibrio fibrisolvens |
| 种 | 蛋白溶解梭菌 | Clostridium proteoclasticum |
| 种 | 金黄色葡萄球菌 | Staphylococcus aureus |
| 种 | 青春双歧杆菌 | Bifidobacterium adolescentis |
| 种 | 鼠李糖乳杆菌 | Lactobacillus rhamnosus |
| 种 | 粪肠球菌 | Enterococcus faecalis |
| 种 | 呀卟啉单胞菌 | Porphyromonas gingivalis |
| 种 | 阴道加德纳菌 | Gardnerella vaginalis |
| 种 | 鼠乳杆菌 | Lactobacillus murinus |
| 种 | 解淀粉芽孢杆菌 | Bacillus amyloliquefaciens |
| 种 | 芽孢杆菌 | Bacillus velezensis |
| 种 | Akkermansia muciniphila | |
| 种 | 酿酒酵母 | Saccharomyces cerevisiae |
| 株 | 唾液链球菌 K12 | Saliva streptococcs |
2 qPCR的原理
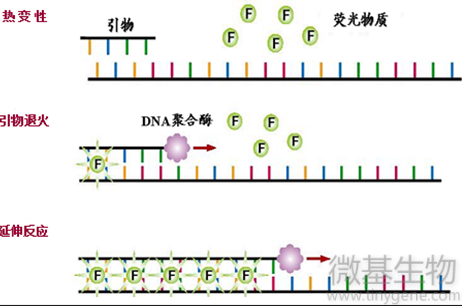
实时荧光定量PCR技术(Real-timeQuantitativePCR,qPCR)是指在PCR反应体系中加入可与DNA产物特异性结合的荧光基团,利用荧光信号积累实时监测整个PCR进程,最终通过相对定量(与内参基因对比)或绝对定量(通过标准曲线对未知模板进行定量)的方法确定各个样本的本底表达量。
3 3种荧光试剂的工作原理及区别
SYBRGreenⅠ法:
嵌入到双链DNA分子后在PCR反应体系中,加入过量SYBR荧光染料,SYBR荧光染料特异性地掺入DNA双链后构象发生变化,能够吸收497nm的激发光并发出520nm的荧光;而不掺入DNA双链中的染料分子不会发射任何荧光信号,从而保证荧光信号的增加与PCR产物的增加完全同步。
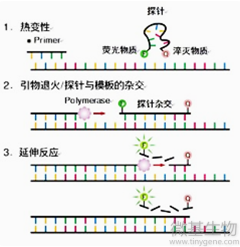
TaqMan探针法:
扩增时加入一个特异性的寡核苷酸荧光探针,两端分别标记一个报告荧光基团和一个淬灭荧光基团。探针完整时,报告基团发射的荧光信号被淬灭基团吸收;PCR扩增时,Taq酶的5′-3’外切酶活性将探针酶切降解,使报告荧光基团和淬灭荧光基团分离,从而荧光监测系统可接收到荧光信号,即每扩增一条DNA链,就有一个荧光分子形成,实现了荧光信号的累积与PCR产物形成完全同步
SYBRGREEN法和TaqMan探针法的区别
| 方法 | 优点 | 缺点 |
|
SYBR GREEN法 |
高灵敏度,操作简单,不影响酶促反应,价格便宜 |
1与探针法相比,对引物特异性要求较高,需进行熔解曲线分析 2灵敏度相对较低。 |
|
TaqMan 探针法 |
1具有高度特异性 2更高的灵敏度 3可同时检测几种不同的荧光信号的产物 |
1探针价格较高 2需要不同引物的扩增效率一致,对引物的设计及扩增条件要求比较高 |
4 qPCR的实验方法
| 实验方法 | 原理 | 技术应用 | 相关应用 |
| 绝对定量(AbsoluteQuantification,AQ) | 是测定目的基因在样本中的分子数目,通过构建标准曲线对未知模板进行分子数目的定量,即通常所说的拷贝数。 |
特定微生物的拷贝数检测 特定功能基因的拷贝数检测 特定抗性基因的拷贝数检测 |
1临床疾病诊断 2动物疾病检测 3食品安全 4科学研究 5应用行业:各级各类医疗机构、大学及研究所、CDC、检验检疫局、兽医站、食品企业及乳品厂等。 |
| 相对定量(RelativeQuantification,RQ) | 测定目的基因在样本中的含量的相对比例,而不需要知道它们在每个样本中的绝对拷贝数,一般是通过CT值之差来计算。 |
样本中某种特定微生物拷贝数占全部微生物拷贝数的比例 某种功能基因的微生物拷贝数占全部微生物拷贝数的比例 某种抗性基因拷贝数占全部微生物拷贝数的比例 |
|
相对定量和绝对定量的数据计算方法
标准质粒拷贝数计算:
每微升拷贝数(copies/μl)=[质粒浓度(ng/ul)×10-9×6.02×1023]/克隆产物相对分子质量
原始样本目标基因拷贝数copies/μLDNA=拷贝数*稀释倍数
原始样本目标基因拷贝数copies/g样本=(原始样本目标基因拷贝数copies/μLDNA*基因组DNA体积)/样本抽提重量,(如果客户样本为DNA样本,则不需要计算copies/g样本)
相对定量的计算:
步骤1:内参基因均一化样本差异:Ct目的基因–Ct内参基因=△Ct
步骤2:其他样本和对照样本比较:△Ct处理样本–△Ct对照样本=△△Ct
步骤3:使用公式计算:倍数变化=2-△△Ct
5 qPCR的实验流程
6 qPCR检测送样要求
| 样本类型 | 样品量/例 | 备注 | |
| 环境 | 土壤/沉积物/淤泥 | 1g | |
| 湖水/海水/河水 | 1L,用滤膜或离心富集 | 过 0.22μm 的滤膜,或者 12000rpm 离心,进行富集。 | |
| 污水 | 20ml | 若样本清亮则可适当地多取。 | |
| 泥水混合样 | 2ml | ||
| 空气 | 根据实验需要,用无菌滤膜过滤空气。 | ||
| 发酵物 | 固体 2g,液体 20ml |
| 样本类型 | 样品量/例 | 备注 | |
| 人体 | 粪便 | ≥3g | |
| 皮肤 | 5 个采集拭子 | 采集 5cm*5cm 面积,反复刮取 20 次。 | |
| 生殖道 | 5 个采集拭子 | 采集阴道口内 4cm 处分泌物, 每个拭子转 3 圈。 | |
| 牙菌斑/舌苔 | 5 个采集拭子 | 在采集的部位,刮取 10 次左右。 | |
| 唾液 | 10ml | ||
| 痰液 | 10ml 或 2-3 口痰液 | ||
| 鼻腔 | 5 个采集拭子 | 采集鼻腔内粘膜上的分泌物,转 3 圈。 | |
| 肺部灌洗液 | 30ml 富集液 | 对灌洗液进行离心富集。 | |
| 肠道/胃组织 |
5mm*5mm*3mm 3 块 |
尽量多一取些。 | |
| 血液 | 3ml 全血 | 用 EDTA 抗凝管,颠倒 8-10 次。 不建议用肝素。 | |
| 尿液 | 30mL 尿液 | 取中段尿为宜。 | |
| 母乳 | 5mL | ||
| 动物 | 粪便 | ≥3g | 最少 0.05g。 |
|
盲肠/结肠/胃 组织 |
≥3g | 至少 1g,如果实验条件允许,尽可能多的收集样本 | |
| 肠道内容物 | ≥3g | 最少 0.05g。建议客户自己取内容物,可以用 PBS 冲洗。 |
提醒
1 因为qPCR绝对定量最终得到的是单位重量,或是单位体积目标基因的拷贝数,所以如果客户送DNA,建议客户记录抽提DNA时样本的使用重量或是体积,方便后继对数据进行转化。
2 送样时请尽量使用冰盒(泡沫盒+干冰),以保证运输过程中的低温条件(干冰的挥发消耗量约为 3-4 公斤/天)。在打包时放入足量的干冰, 将样本埋入干冰中, 为了保持冰盒中的低温环境,建议采用加厚的泡沫盒
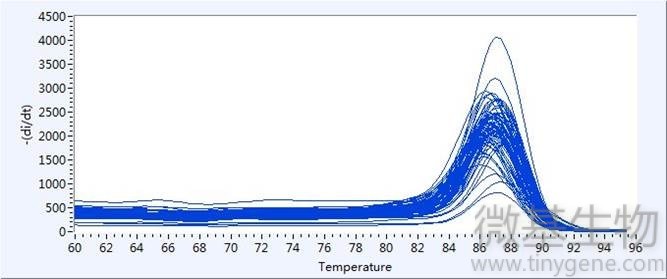
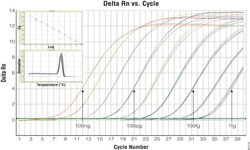
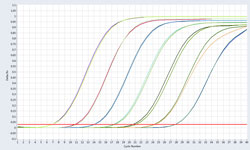
7 qPCR结果
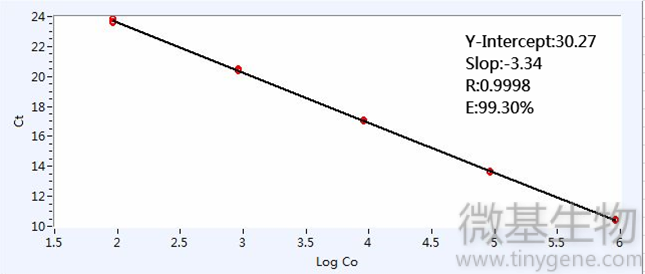
标准曲线
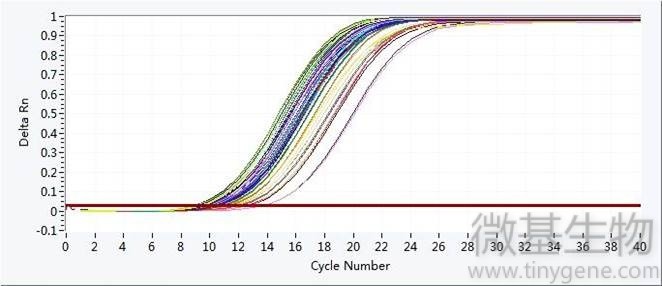
扩增曲线
熔解曲线
定量结果
| 微基编号 | 拷贝数 | 样本稀释倍数 | 抽提样本重量(g) | 基因组DNA体积(μL) | 原始样本目标基因拷贝数copies/μL DNA | 原始样本目标基因平均拷贝数copies/μL DNA | 原始样本目标基因拷贝数copies/g样本 | 原始样本目标基因平均拷贝数copies/g样本 |
| 1 | 1.08E+06 | 200 | 0.237 | 50 | 2.17E+08 | 2.26E+08 | 4.58E+10 | 4.76E+10 |
| 1 | 1.23E+06 | 200 | 0.237 | 50 | 2.47E+08 | 5.20E+10 | ||
| 1 | 1.07E+06 | 200 | 0.237 | 50 | 2.14E+08 | 4.51E+10 | ||
| 2 | 7.28E+05 | 200 | 0.24 | 50 | 1.46E+08 | 1.49E+08 | 3.03E+10 | 3.09E+10 |
| 2 | 7.55E+05 | 200 | 0.24 | 50 | 1.51E+08 | 3.15E+10 | ||
| 2 | 7.44E+05 | 200 | 0.24 | 50 | 1.49E+08 | 3.10E+10 | ||
| 3 | 1.24E+06 | 200 | 0.25 | 50 | 2.48E+08 | 2.59E+08 | 4.96E+10 | 5.19E+10 |
| 3 | 1.35E+06 | 200 | 0.25 | 50 | 2.69E+08 | 5.39E+10 | ||
| 3 | 1.30E+06 | 200 | 0.25 | 50 | 2.61E+08 | 5.21E+10 | ||
| 4 | 1.61E+06 | 200 | 0.234 | 50 | 3.21E+08 | 3.22E+08 | 6.87E+10 | 6.87E+10 |
| 4 | 1.60E+06 | 200 | 0.234 | 50 | 3.19E+08 | 6.82E+10 | ||
| 4 | 1.62E+06 | 200 | 0.234 | 50 | 3.24E+08 | 6.93E+10 | ||
| 5 | 1.27E+05 | 200 | 0.175 | 50 | 2.54E+07 | 2.66E+07 | 7.25E+09 | 7.59E+09 |
| 5 | 1.33E+05 | 200 | 0.175 | 50 | 2.66E+07 | 7.61E+09 | ||
| 5 | 1.39E+05 | 200 | 0.175 | 50 | 2.77E+07 | 7.91E+09 |
8 术语解释
Ct值(Cyclethreshold,循环阈值):每个反应管内的荧光信号到达设定阈值时所经历的循环数。
荧光阈值(threshold)的设定:PCR反应的前15个循环的荧光信号作为荧光本底信号,荧光阈值的缺省(默认)设置是3-15个循环的荧光信号的标准偏差的10倍,即:threshold=10*SDcycle3-15
基线(baseline):在PCR扩增反应的最初数个循环里,荧光信号变化不大,接近一条直线,即称为基线。
熔解曲线Tm(TmOfMeltingCurve):SYBRGreen染料发实验结束后需对qPCR产物加热,随着温度的升高,双链接扩增产物逐渐解链,导致荧光强度下降,到达某一温度时,会导致大量的产物解链,荧光急剧下降,将此温度称为Tm值。不同PCR产物Tm值不同,从而可对PCR的特异性进行鉴定。
9 参考文献
1 Tristano Bacchetti De Gregoris et.al. Improvement of phylum- and class-specific primers for real-time PCR quantification of bacterial taxa.2011. Journal of Microbiological Methods.(厚壁菌门拟杆菌门alpha变形菌gamma变形菌等)
2 Baskar Balakrishnan et.al. Development of a real-time PCR method for quantification of Prevotella histicola from the gut.2017.Anaerobe.(普氏菌)
3 John Penders et.al. Quantification of Bifidobacterium spp., Escherichia coli and Clostridium difficile in faecal samples of breast-fed and formula-fed infants by real-time PCR.2005. FEMS Microbiology Letters.(双歧杆菌艰难梭菌大肠杆菌探针法)
4 Anders Bergstrom et.al. Introducing GUt Low-Density Array (GULDA) – a validated approach for qPCR-based intestinal microbial community analysis.2012. FEMS Microbiology Letters.(多种菌的定量)

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用