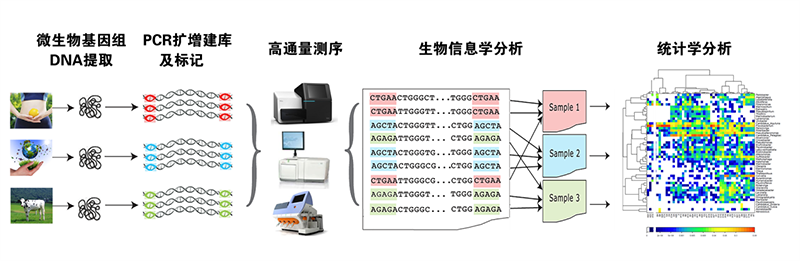
微基生物提供土壤微生物多样性分析的整体科研服务:实验规划->样本采集保存->分子实验->生信统计分析->论文协助
微基生物采用高通量测序、PCR-DGGE、实时荧光定量PCR等方法,对样本中的DNA进行序列测定,并通过生信统计分析,对大量数据进行处理,揭示肠道中微生物的种类以及它们之间的相对丰度和进化关系,探讨微生物多样性,研究土壤微生物与环境间的相关关系。
技术路线:
高通量分析流程
PCR-DGGE分析流程
检测平台:
微基生物拥有Illumina MiSeq、Ion PGM、Roche 454高通量测序分析,PacBio第三代高通量测序分析,PCR-DGGE变性梯度凝胶分析,实时荧光定量PCR(Real-time qPCR),克隆文库等检测平台。
样品采集:
微基生物为客户提供样品采集的配套工具,如采集盒、保存液、取样勺和保存管等。
送样要求:
样品原样
(1)样品类型:土壤,新鲜取样,冻存于-80℃
(2)样品需求:≥2g
(3)样品保存期间切忌反复冻融,送样时请使用冰袋或干冰运输
DNA类型
(1) 样品类型: DNA
(2) 样品需求量:≥300ng
(3) 样品浓度: ≥10ng/μL
(4) 样品纯度:OD260/280=1.8-2.0并确保DNA无降解
(5) 样品保存期间切忌反复冻融,送样时请使用冰袋或干冰运输
(6) 对于本种类型的样品,我们在检测完样品的质量后,进行PCR扩增等后续试验
生物信息与统计学服务:
生信分析项目
更多微生态方向研究和生物信息方面服务,请详询:400-660-9270
案例分析
标题:由长期的土壤移植引起的纬度和气候变化明显改变了土壤微生物的变化率
研究领域:土壤微生物
分析物种:细菌
取样方法:从中科院封丘站取1.4*1.2*1.0体积的土壤,分别向北移植到黑龙江海伦站,向南移植到江西鹰潭站。每组设3个重复,于2006-2011年每年的8-9月取20 cm的表层土,密封在聚乙烯包装袋中,于-80︒C保存。
高通量测序平台:Illumina MiSeq 2×150
测序区域:16S rRNA gene V4区
样本数及分组:分3组,3个重复,共63个样本。
研究背景:
生物群体对由某些人为因素造成的潜在威胁(如气候改变)的响应是目前生态学研究的一个重要挑战。鉴于微生物在生物地球化学循环中的重要作用,它们对气候改变的响应有可能导致生态结构的改变。之前已有研究指出,温度是影响土壤微生物组成和生态功能(土壤的呼吸作用、有机物的含量、固氮水平)的重要因素。而不同地域之间的土壤移植为研究微生物群落对气候变化的响应提供了新的研究方法和思路。
[/tabs]
主要结果:
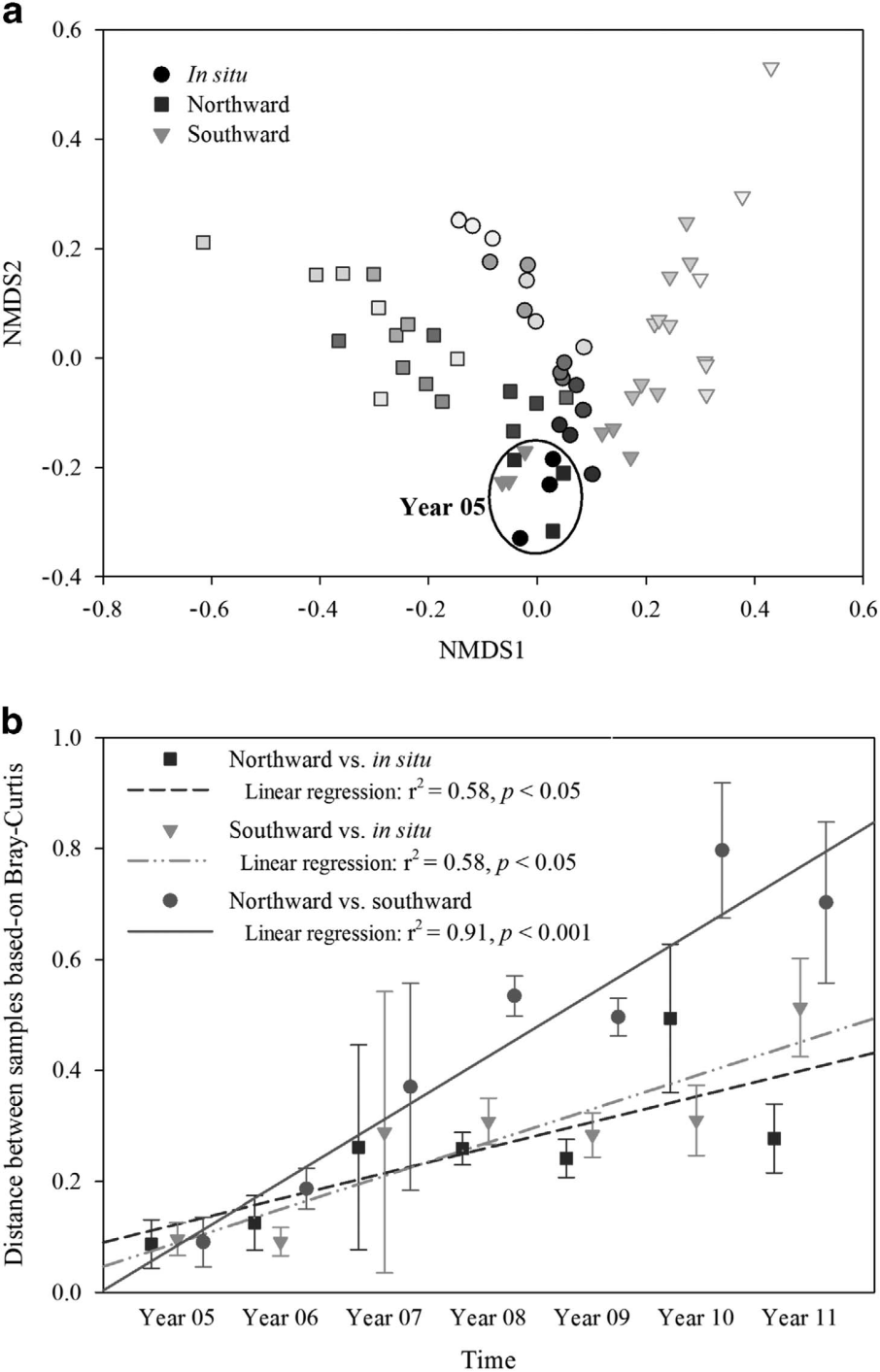
(1)微生物的演替
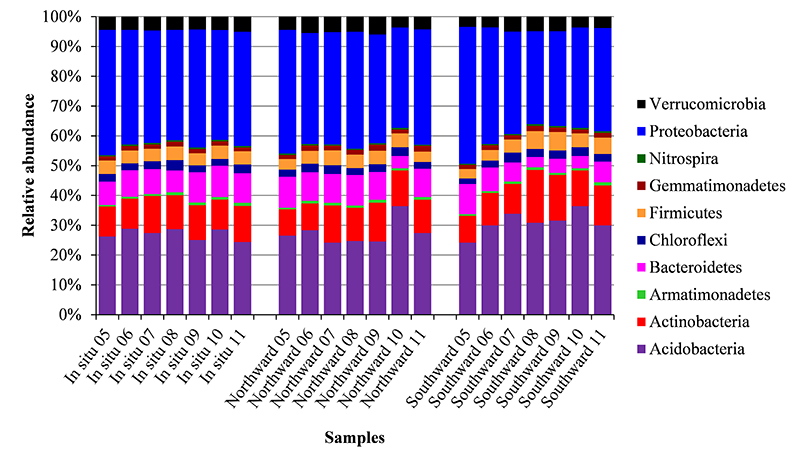
随着时间的推移,向北、向南移植的土壤中,微生物的群落组成差异越来越大,微生物多样性逐渐增加。
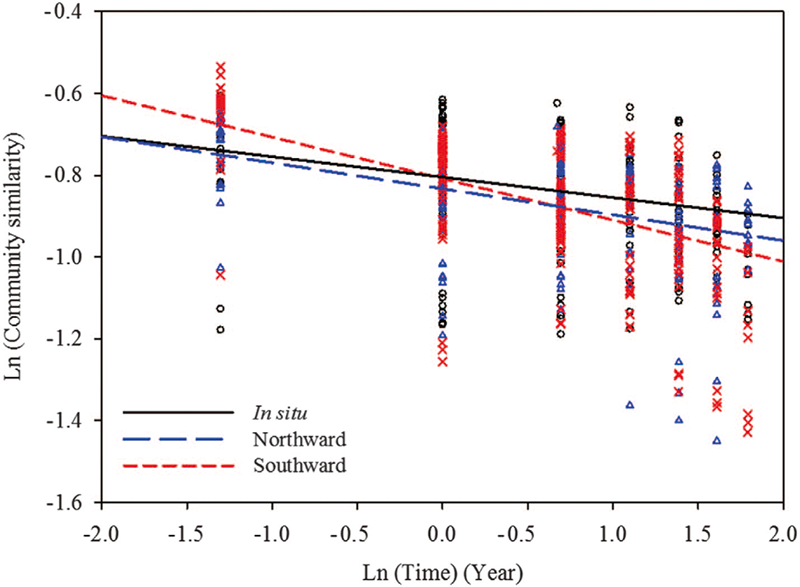
(2)由土壤移植引起的土壤微生物的变化
微生物随时间衰减的斜率可以用来衡量微生物群落的相似性。本研究中,在各个样本的微生物群落中都存在显著的的时间衰减关系。向北、向南移植的土壤中,微生物随时间衰减的斜率均比原位土壤的斜率大,尤其是在向南移植的土壤中。
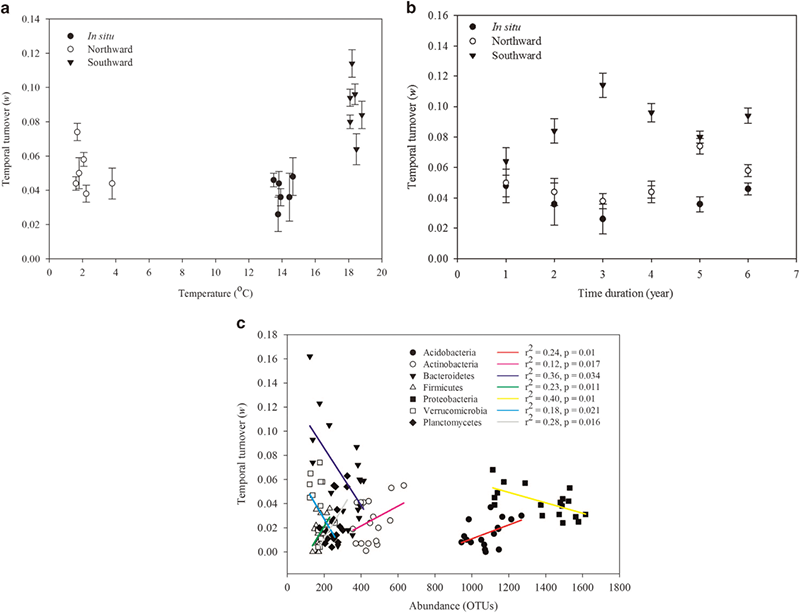
随着温度的升高/降低,微生物的变化率也相应地增加。随着土壤向南移植,气候变暖,微生物群落的波动性增大,微生物群落的变化增加。向南移植对土壤微生物群落的变化具有出更好的效果。有趣的是,研究还发现细菌群落(门水平)的改变与分类学的分度有关。其中变形菌门、拟杆菌门和疣微菌门与门的丰度呈负相关,而酸杆菌门、放线菌门、后壁菌门和浮霉菌门与门的丰度呈正相关。
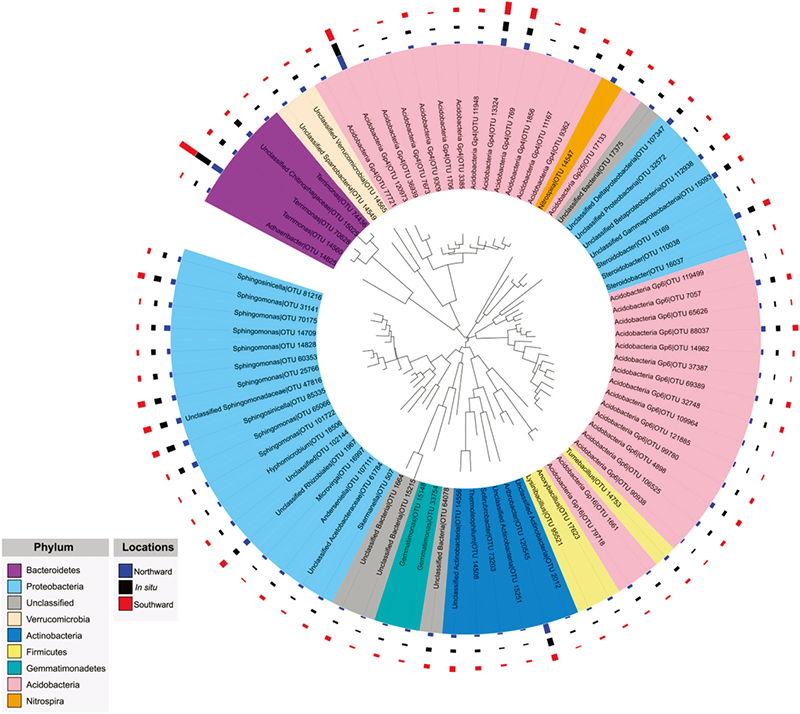
在为期六年的试验中,三个样本共检测出78个OTUs,其OTU的数量非常少,分属于9个门。用MEGA 5作系统进化树,如下图所示。
其中,酸杆菌门中的Gp4和Gp6、节细菌属、硝化螺菌属、鞘氨醇单胞菌、Fervidicoccus、Sphingosinicella、Steroidobacter和Terrimonas是主要菌群。
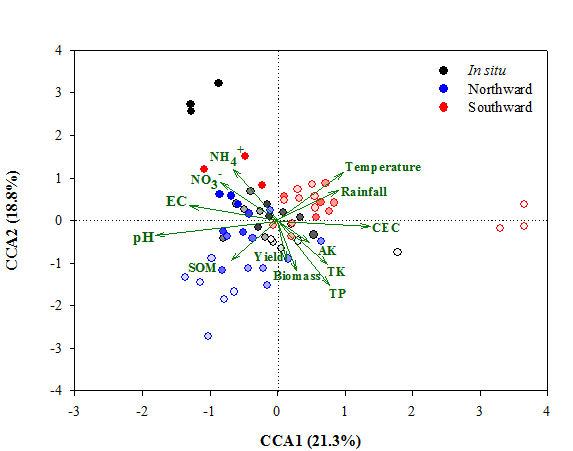
微生物的演替与环境因子之间的关系用CCA分析如下。结果表明,向北移植的土壤中,微生物群落的多样性与土壤的物理化学因子有关;而向南移植的土壤中,微生物群落的多样性受温度和降水的影响较大。
本研究采用illumina MiSeq测序平台,对经过移植的土壤微生物为研究对象,扩增了细菌16S rRNA基因的V4区域,从而得到土壤中细菌种群分布的相关信息,对其中的微生物种群变化进行了调查研究。
此实验采用illumina MiSeq 2*150双端测序,每个样品可得到948,765条序列,其中有效序列为10,947条。研究发现,与原位土壤相比,向北移植(低温)的土壤中,细菌的丰度增加,演替速率升高;向南移植(高温)的土壤中,细菌的丰度降低,而演替速率达到最高,即稳定性不好。推断这是由于高温环境容易引起高的代谢率和更加激烈的生存竞争造成的。
原文链接:http://www.nature.com/ismej/journal/vaop/ncurrent/full/ismej201578a.html
参考文献:
Liang, Y., Y. Jiang, F. Wang, C. Wen, Y. Deng, K. Xue, Y. Qin, Y. Yang, L. Wu, J. Zhou and B. Sun (2015). “Long-term soil transplant simulating climate change with latitude significantly alters microbial temporal turnover.” ISME J.
微基生物进行微生态与环境相关的研究与应用:

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用