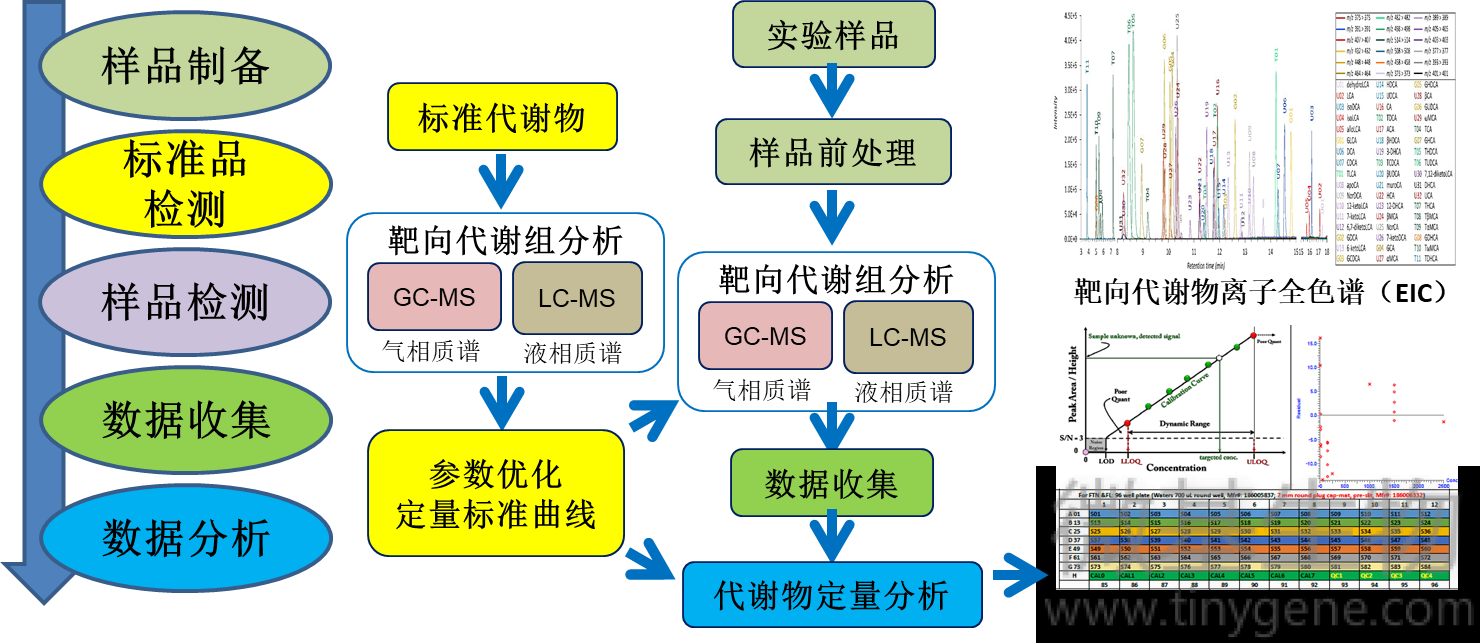
靶向代谢组学是对特定的代谢物群进行检测与分析。分析时加入了标准品的定量分析,用以对特定代谢物绝对浓度的定量检测。常规分析法有气质联用(GC-MS)和液质联用(LC-MS),我们优先利用LC-MS的方法进行代谢组分析[1],然后通过与相同分析条件下标准品的分析结果进行比对,最终完成目标代谢群定性和定量的分析。
针对不同靶向检测物,我们制备了多种标准品,可对 胆汁酸、短链脂肪酸、TMAO及相关代谢物、 氨基酸、脂肪酸、有机酸、黄酮类、 植物激素、 神经递质等进行靶向检测。
实验方法
结果展示
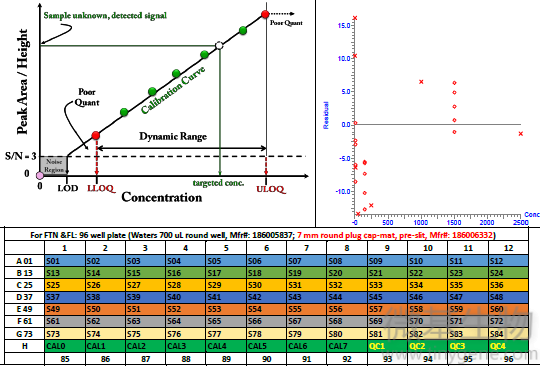
校准曲线和质量控制偏差
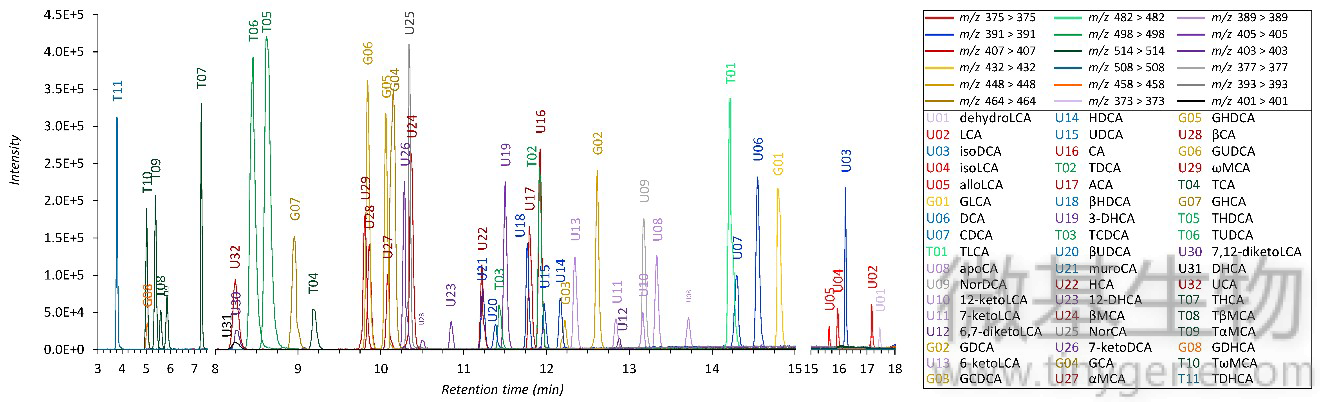
所有单个胆汁酸的提取离子色谱(EIC、全谱)
| HS1B | HS2B | HS3B | HS1C | HS2C | HS3C | |
| TUDCA | 3.76 | 3.7 | 2.96 | 4.5 | 4.42 | 3.26 |
| TLCA | 0.335 | 0.338 | 0.361 | 0.233 | 0.185 | 0.166 |
| GHDCA | 43.2 | 41.3 | 41 | 56.1 | 50.2 | 40.7 |
| λ-GMCA | 2.46 | 2.49 | 2.69 | 3.25 | 3.06 | 3.08 |
| β-MCA | 0.545 | 0.661 | 0.142 | 0.112 | 0.156 | 0.0744 |
| DLCA | 0.616 | 0.439 | 0.417 | 0.2 | 0.154 | 0.0961 |
| NCA | 3.04 | 2.68 | 2.61 | 3.56 | 3.35 | 3.17 |
| 7-KDCA | 1.39 | 1.42 | 1.34 | 1.44 | 1.67 | 1.35 |
| NDCA | 0.195 | 0.261 | 0.162 | 0.409 | 0.302 | 0.315 |
| GLCA | 1.69 | 1.39 | 1.43 | 1.33 | 1.16 | 1.2 |
| β-HDCA | 0.817 | 1.22 | 1.17 | 0.672 | 0.37 | 0.192 |
| UDCA | 30 | 27.7 | 29.2 | 32 | 28 | 24.1 |
| CDCA | 69 | 64.4 | 71.1 | 89.5 | 83.8 | 67.4 |
| DCA | 32.4 | 29.3 | 30.2 | 35.6 | 34.8 | 27.3 |
| THDCA | 2.61 | 1.75 | 1.17 | 3.81 | 2.5 | 2.04 |
| TDCA | 10.2 | 9.23 | 8.99 | 12.8 | 10.7 | 10.1 |
| GUDCA | 8.37 | 6.75 | 7.05 | 10.9 | 8.79 | 7.6 |
| GCDCA | 285 | 260 | 256 | 364 | 314 | 267 |
| GDCA | 48 | 47.1 | 50.3 | 63.8 | 54.2 | 44.3 |
| GCA | 129 | 118 | 125 | 142 | 137 | 135 |
| UCA | 0.917 | 0.592 | 0.864 | 1.18 | 1.15 | 1.07 |
| 3β-CA | 1.78 | 2.22 | 0.923 | 1.56 | 1.43 | 1.32 |
| α-MCA | 0.214 | 0.607 | 0.245 | 0.152 | 0.127 | 0.193 |
| λ-MCA | 2.08 | 2.19 | 2.16 | 3.93 | 3.34 | 2.61 |
| CA | 83.3 | 56.4 | 52.5 | 54 | 51.9 | 46 |
| ALCA | 0.434 | 1.05 | 0.654 | 0.587 | 0.677 | 0.637 |
| ω-TMCA | 0.463 | 0.434 | 0.4 | 0.795 | 0.435 | 0.542 |
| TCA | 27 | 26 | 24.6 | 35.2 | 30.5 | 27.4 |
| 7-KLCA | 2.16 | 2.2 | 2.49 | 1.5 | 1.9 | 1.38 |
| APCA | 0.475 | 0.604 | 0.432 | 0.416 | 0.613 | 0.294 |
| 3-DHCA | 0.491 | 0.336 | 0.525 | 0.491 | 0.336 | 0.525 |
| 12-DHCA | 8.77 | 8.87 | 8.47 | 9.67 | 12.6 | 8.19 |
| TCDCA | 60.24 | 59.3 | 57.33 | 36.45 | 35.65 | 39.22 |
胆汁酸检测结果
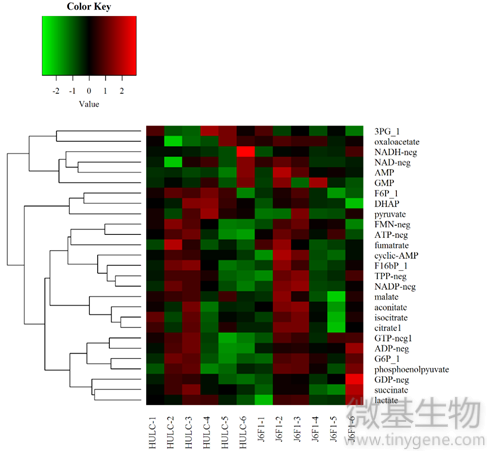
代谢物通路分析
参考文献
1 在基于LC-MS的整体代谢组分析过程中,我们所遇到的问题、局限、优势和未来展望。 Gika H.G., et al. 2014. LC-MS-based holistic metabolic profiling. Problems, limitations, advantages, and future perspectives. J Chromatogr B Analyt Technol Biomed Life Sci. 2014 Sep 1;966:1-6. doi: 10.1016/j.jchromb.2014.01.054. Epub 2014 Feb 8.
2 代谢组学在人体肠道微生态研究中的机遇和挑战。Smirnov K.S., et al. 2016. Challenges of metabolomics in human gut microbiota research. Int J Med Microbiol. 2016 Aug;306(5):266-279. doi: 10.1016/j.ijmm.2016.03.006. Epub 2016 Mar 15.

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用