ITS序列是内源转录间隔区(Internally Transcribed Spacer),位于真菌18S、5.8S和28S rRNA基因之间,分别为ITS 1和ITS 2。 在真菌中,5.8S、18S和28S rRNA基因具有较高的保守性,而ITS由于承受较小的自然选择压力,在进化过程中能够容忍更多的变异,在绝大多数真核生物中表现出极为广泛的序列多态性。同时,ITS的保守型表现为种内相对一致,种间差异较明显,能够反映出种属间,甚至菌株间的差异。并且ITS序列片段较小(ITS 1和ITS 2长度分别为350 bp和400 bp),易于分析,目前已被广泛用于真菌不同种属的系统发育分析。 微基生物进行人体健康相关的研究与应用:
微基生物提供与人体健康相关的生信/统计服务: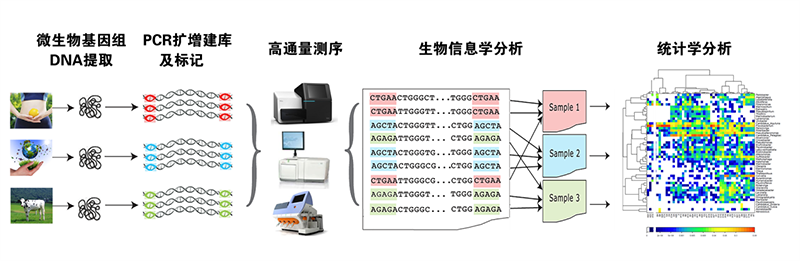
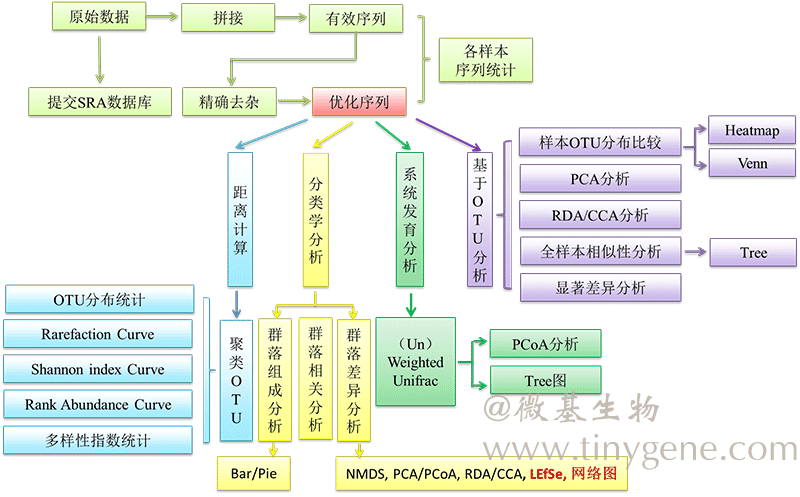
目前市面上常用的用于研究环境微生物的高通量测序平台有Illumina MiSeq,Roche 454和Ion Torrent PGM。针对于真菌ITS序列,MiSeq凭借其测序读长长、测序周期短、通量大等特点,成为使用最为普遍的测序平台。 微基生物是国内首家采用Illumina-MiSeq 2×300 bp平台进行微生物生态研究,其最大读长可达到550 bp,能够很好地覆盖ITS 1和ITS 2序列,对其进行测序分析。
案例分析
标题:Integrated Meta-omics Approaches To Understand the Microbiome of Spontaneous Fermentation of Traditional Chinese Pu-erh TeamSystems (IF: 6.633)
研究领域:食品
分析物种:细菌,真菌
高通量测序平台:Illumina MiSeq
测序区域:V4V5,ITS 1
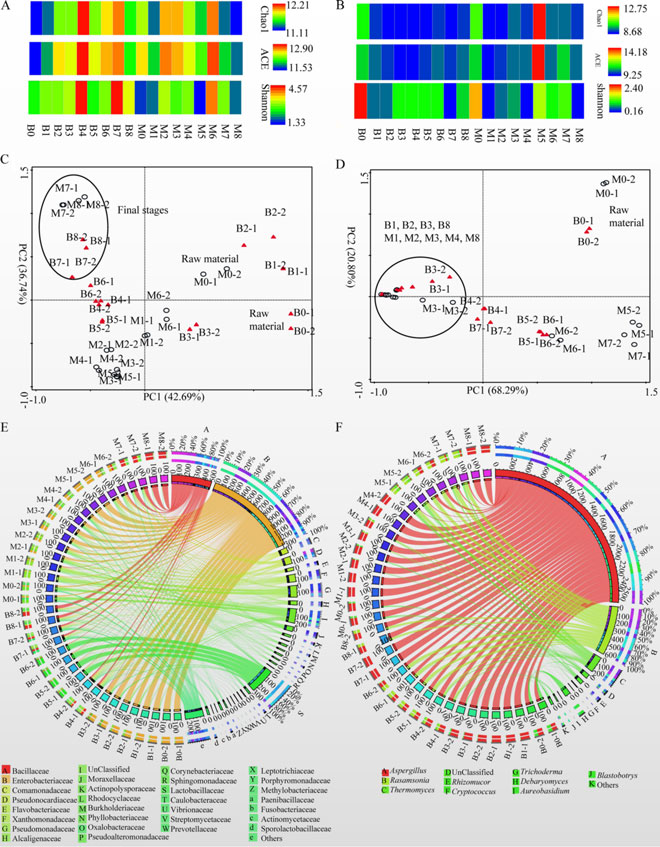
主要结果:
原文链接:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6867877/
参考文献:Zhao M, Su XQ, Nian B, et al. Integrated Meta-omics Approaches To Understand the Microbiome of Spontaneous Fermentation of Traditional Chinese Pu-erh Tea. mSystems. 2019;4(6):e00680-19. Published 2019 Nov 19. doi:10.1128/mSystems.00680-19

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用