LEfSe是一种用于发现高维生物标识和揭示基因组特征的软件。包括基因,代谢和分类,用于区别两个或两个以上生物条件(或者是类群)。该算法强调的是统计意义和生物相关性。让研究人员能够识别不同丰度的特征以及相关联的类别。
LEfSe 通过生物学统计差异使其具有强大的识别功能。然后,它执行额外的测试,以评估这些差异是否符合预期的生物学行为。具体来说,首先使用non-parametric factorial Kruskal-Wallis (KW) sum-rank test(非参数因子克鲁斯卡尔—沃利斯和秩验检)检测具有显著丰度差异特征,并找到与丰度有显著性差异的类群。最后,LEfSe采用线性判别分析(LDA)来估算每个组分(物种)丰度对差异效果影响的大小。
LDA:使用这种方法能够使投影后模式样本的类间散布矩阵最大,并且同时类内散布矩阵最小。就是说,它能够保证投影后模式样本在新的空间中有最小的类内距离和最大的类间距离,即模式在该空间中有最佳的可分离性。
分析软件:LEfSE[2](http://huttenhower.sph.harvard.edu/galaxy/root?tool_id=lefse_upload)根据分类学组成对样品按照不同的分组条件进行线型判别分析(LDA),找出对样品划分产生显著性差异影响的群落或物种。
参考文献:
[1] Chenhong Zhang, Shoufeng Li, Liu Yang, et al. Structural modulation of gut microbiota in life-long calorie-restricted mice. NATURE COMMUNICATIONS, 4:2163, DOI:10.1038/ncomms 3163(2013).
[2] Segata N, Izard J, Waldron L, Gevers D, Miropolsky L et al. (2011) Metagenomic biomarker discovery and explanation. Genome Biol 12: R60.10.1186/gb-2011-12-6-r60 PubMed: 21702898.
例图:
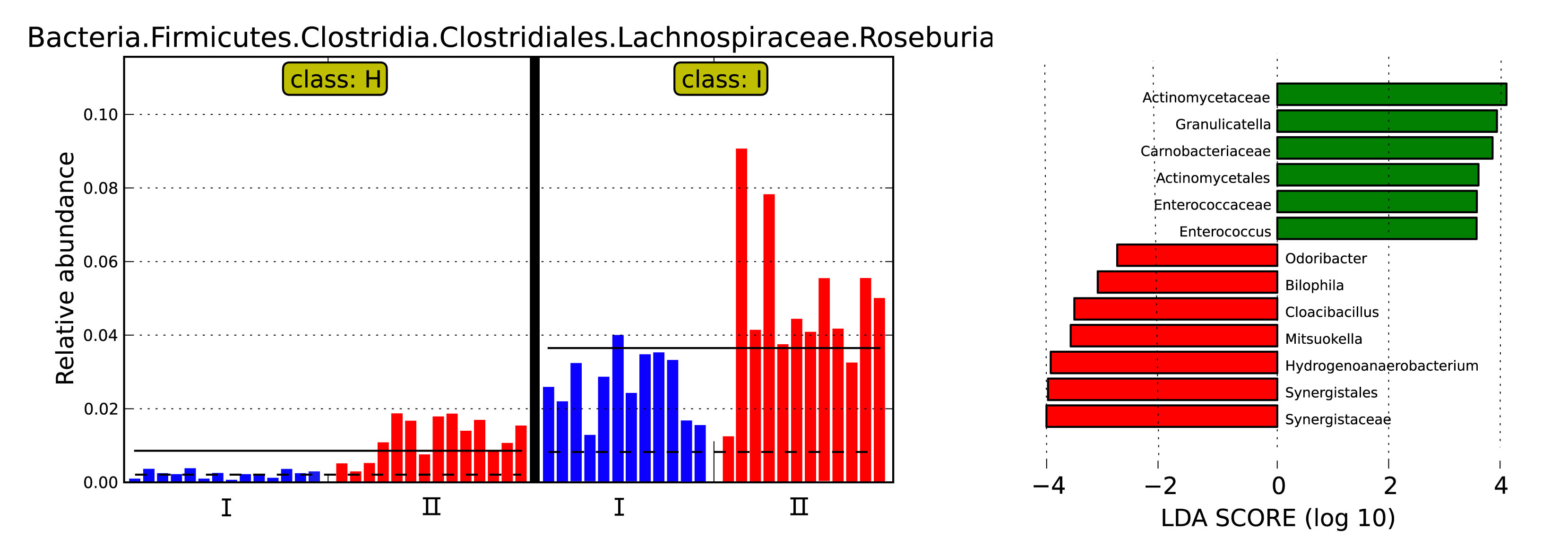
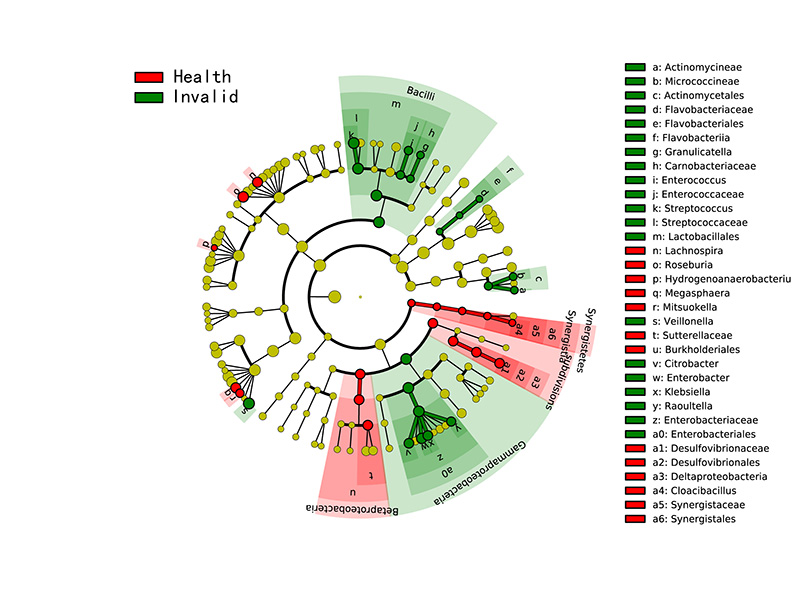
注:左图为聚类树,红色区域和绿色区域表示不同分组,树枝中红色节点表示在红色组别中起到重要作用的微生物类群,绿色节点表示在绿色组别中起到重要作用的微生物类群,黄色节点表示的是在两组中均没有起到重要作用的微生物类群。图中英文字母表示的物种名称在右侧图例中进行展示。右图为统计两个组别当中有显著作用的微生物类群通过LDA 分析(线性回归分析)后获得的LDA 分值。

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用