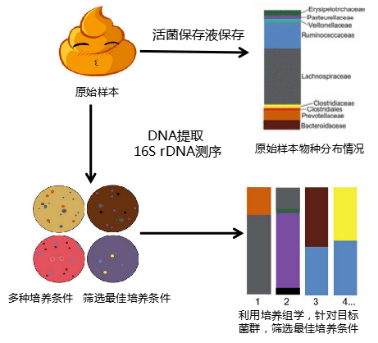
服务简介:利用培养组学(多达48或96种条件)将原始样本中可培养微生物大大富集并进行微生物组学的检测,使原始样本中可检测的微生物类型大为丰富。为此后的微生物的培养分离验证了条件。
使用需氧/厌氧活菌保存液保存样本,微生物的种类更丰富,多样性更显著。
引言
培养组学(Culturomics)是一种综合方法,使用多种培养条件在不同培养基上培养细菌菌落,然后使用16S rDNA测序或MALDI-TOF质谱平台对其进行鉴定,是了解特定菌或菌群在宿主中作用的关键。可以较大程度的发现潜在的新菌株和微生物,丰富现有的可培养微生物资源库。
16S rRNA基因测序或宏基因组测序,极大丰富了微生物组数据库。但测序技术的检测阈值较高,即低丰度细菌可能无法通过测序检出。不能了解具体菌株的实际功能和生理特性。而培养组学通过培养基的设计及培养条件的多样化,可以培养和检测丰度较低的细菌。分离培养出更多细菌类别,对于在菌株水平的研究和推广应用意义重大。因此测序技术与培养组学之间是互补的。
通过多种培养基条件培养与16S rRNA基因测序相结合来检测细菌的多样性,对于研究微生物群更为全面。用于特定菌株的分离培养,从微生物群中分离出感兴趣的细菌,为探索微生物群与宿主相互作用的机制提供了新的机会。
服务内容
1.原始样本二代高通量测序,检测样本中微生物种类及其相对丰度。
2.将样本在实验设定的多种培养基和培养条件中培养,通过二代测序检测物种丰富度。
3.原始样本与培养组菌落测序结果对比,了解细菌的种类及丰富度,是否可培养等情况。
4.根据测序得到物种丰富度选择适合目标菌株生长的培养基及培养条件。
5.后继建立适合特定目标菌株生长的特定培养基及培养条件,分离纯化得到目标菌株。
(参考微基官网:特定菌株代测分离培养https://www.tinygene.com/culture-isolation/special-bacterial-strains)
样本类型
粪便、肠道组织、肿瘤组织、土壤、淤泥、植物、药品等样本中的微生物
名词解释
1.特定菌株的分离培养:利用特定菌的培养基和培养条件,对目的菌进行培养,同时利用96孔板进行大规模的分离、纯化等反复操作,找到目的菌株。对最终纯化好的目的菌株进行菌种鉴定,从基因水平上再次确认目标菌株。
2.16S rRNA测序研究到了属水平,全基因组测序研究到了种的水平,而培养组学深入到株的水平。因此,培养组学的研究深度优于16S rRNA测序和宏基因组学。
文献解读
1.猪肠道微生物的培养
Comprehensive Cultivation of the Swine Gut Microbiome Reveals High Bacterial Diversity and Guides Bacterial Isolation in Pigs(2021)
背景
1.使用53种细菌培养方法,用不同的培养基和气体组合,从三头处于四个不同生长阶段的猪中培养了猪的肠道微生物群。
2.依赖于培养(CD;来自每种方法的菌落混合物)和培养非依赖性(CI;原始粪便悬浮液)样品进行分别进行16S rRNA基因扩增子测序。
3.在连续生长阶段的CI和CD样品中观察到微生物多样性增加。在CI样本中观察到总共378、482、565和555个细菌扩增子序列变异体(asv),使用CD方法分别在泌乳、保育、生长和育肥阶段检测到更高的微生物多样性(415、675、808和823个观察到的asv)。
4.基于培养依赖的方法,猪肠道微生物群比以前所知的更加多样化。
主要结果
(1)猪肠道微生物群在四个生长阶段的非培养(CI)和培养依赖(CD)视图。
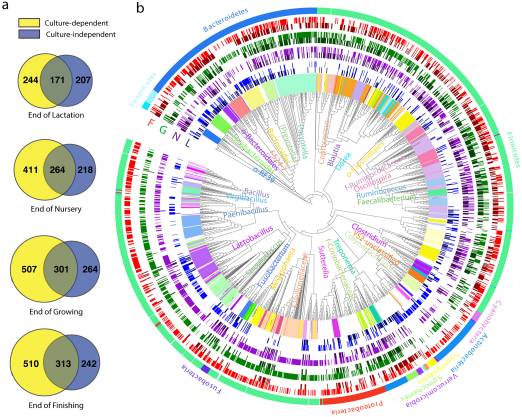
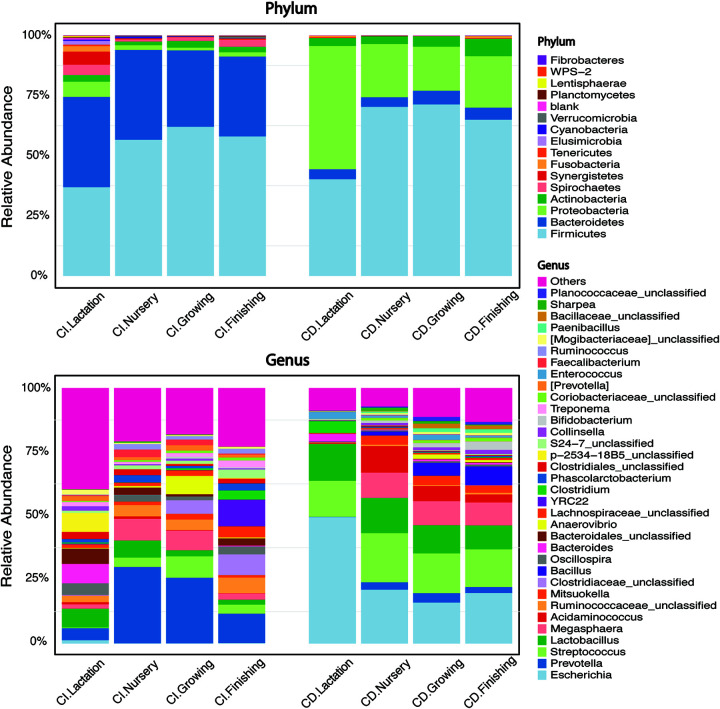
(2)两种培养方法在四个时间点通过16S rRNA基因扩增子测序收集的猪粪中的门和前20个属的堆积条形图。
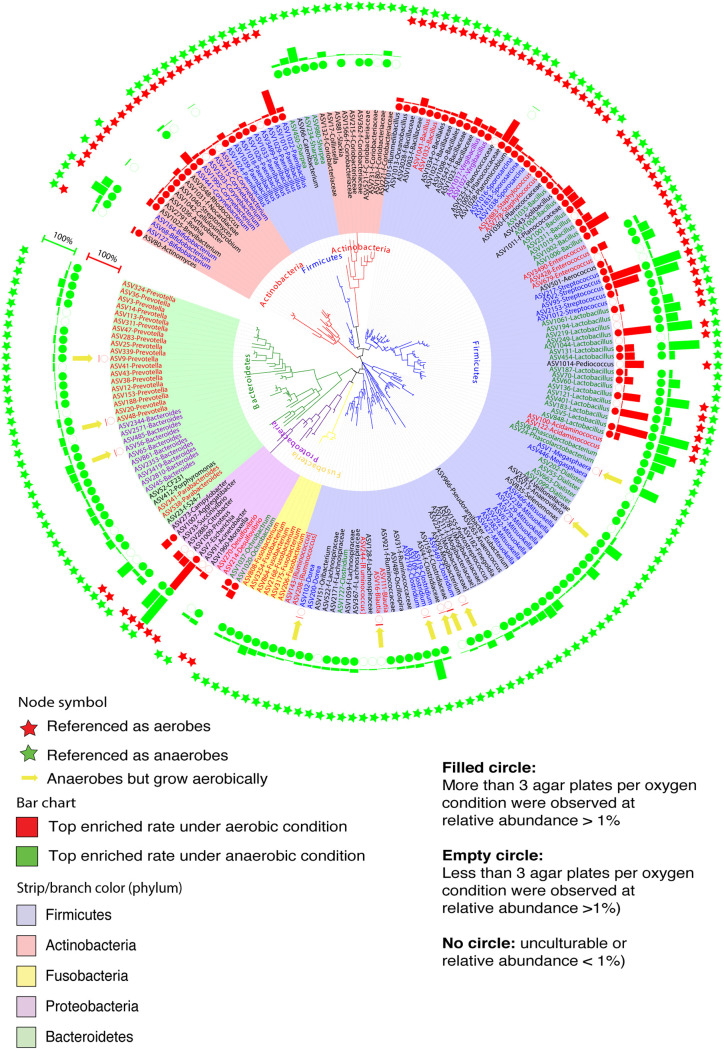
(3)53 种培养方法下(A红色,好氧条件;N蓝色,厌氧条件),猪体内感兴趣的富集特定细菌ASV的热图。

(4)系统进化树分析显示了在需氧和厌氧条件下琼脂平板上检测到的前200个细菌asv。
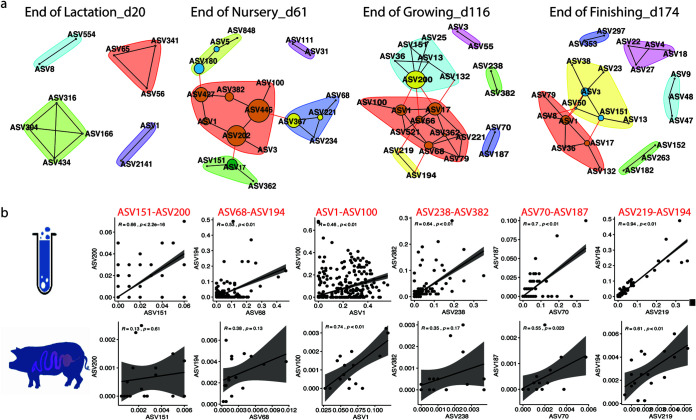
(5)由SparCC(R.0.4,P,0.05)在每个时间点(a)在文化独立(CI)和文化依赖(CD)模型中公开的共享共现网络。
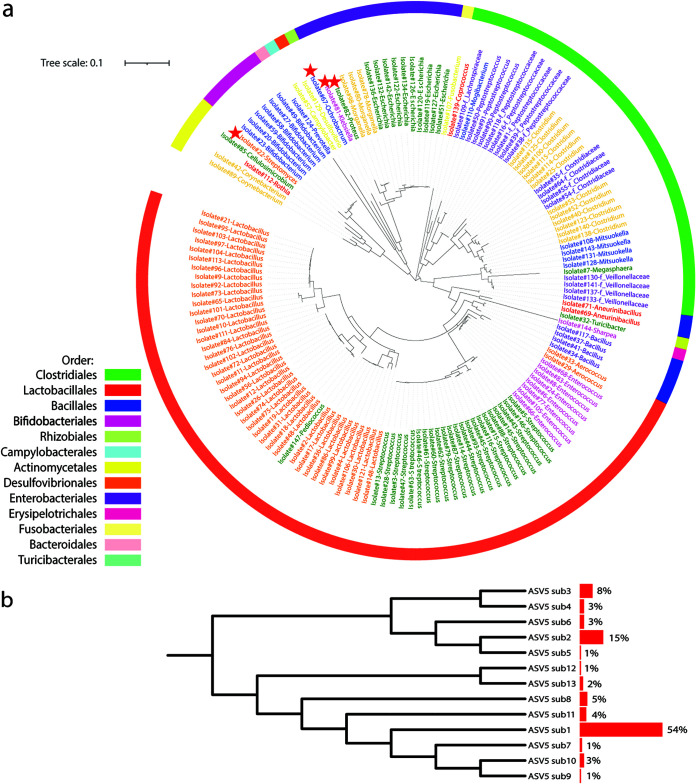
(6)基于16S rRNA基因V3-V7高变区的148种不同细菌分类群的系统发生树分析。
结论
1.本研究采用53种培养方法,对三头不同生长阶段的猪的12份粪便样品进行了研究,以探讨猪肠道微生物的可培养性,并为这方面的研究提供指导。
2.我们的数据显示大约50%的asv和75%的属和科是可培养的。综合培养与不依赖培养的测序方法一起,显著扩展了我们对猪肠道微生物丰富性的认识。我们还分离了许多重要的细菌asv,包括几个新的物种和重要的核心asv,它们被非培养方法所忽略。
3.此外,我们还生成了一个参考热图,用于指导特定目标菌群的培养,例如猪体内的某些益生菌和优势菌群。我们还研究了不同因素如氧气、生长培养基、供体年龄、抗生素和血瓶培养预富集对猪肠道微生物群培养的影响。通过将我们的结果与人类培养组学进行比较,观察到了相似的模式,这加强了猪和人类肠道群落之间相似性的想法。
4.最后,网络分析显示了独立于培养的微生物群之间的共享共现模式,验证了利用体内数据开发的生态模型的发现。
2.人类肠道微生物的培养
Capturing the diversity of the human gut microbiota through culture-enriched
molecular profiling(2016)
背景
1.培养组学和16S rRNA基因测序相结合,可以培养人类肠道微生物群16S测序鉴定的大多数细菌。
2.33种培养基培养5个新鲜的粪便样本,厌氧和需氧培养板产生66种培养条件。
3.粪便微生物群的可培养部分是通过将培养板的16S测序回收的操作分类单位(OTU)与粪便样本的非培养独立测序的OTU进行比较来确定的。
4.两个新鲜粪便样本进行毛螺菌科菌株的靶向分离。
主要结果
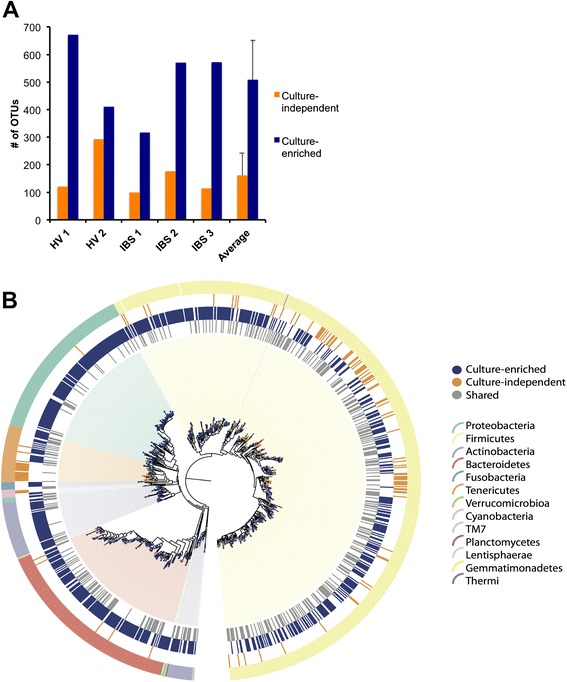
(1)富含培养物的分子谱分析捕获了大部分OTUs。
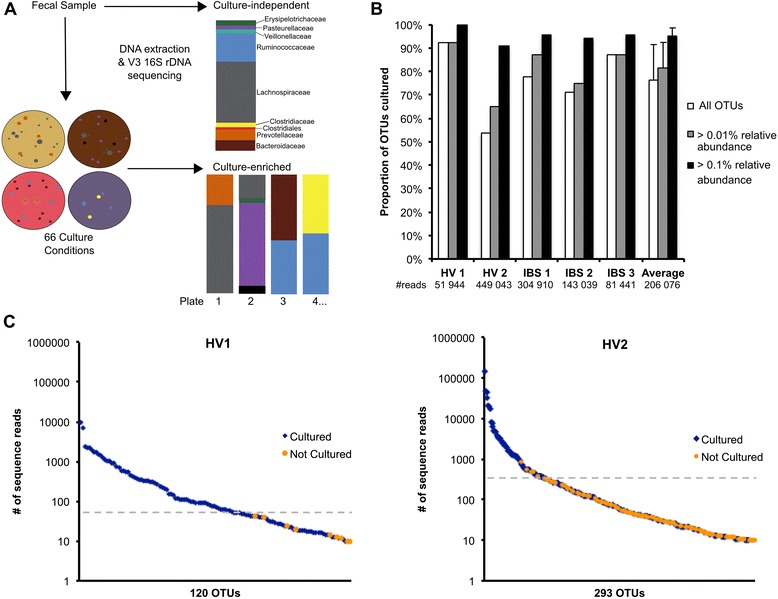
(2)与不依赖于培养物的测序相比,富含培养物的分子谱分析检测到更多的OTU。
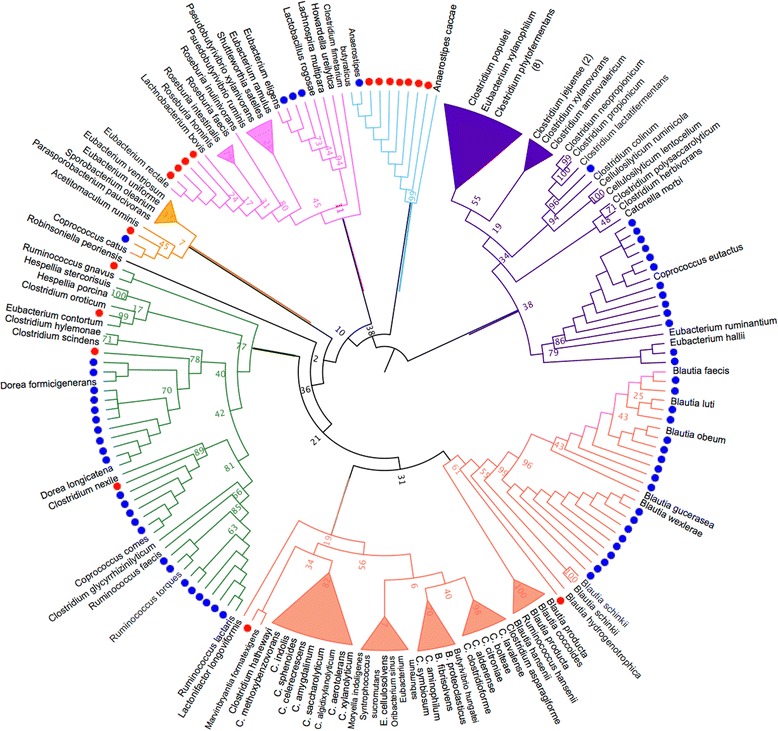
(3) 毛螺菌科分离鉴定:根据之前的营养基中培养测序的结果。新鲜收集的HV7和IBS4粪便样本在BHI+ 1g/L菊粉(BHI+inu)和熟肉琼脂(BEEF)上进行厌氧培养。将分离株的16S rRNA基因序列与人类微生物组计划(HMP)列表中的毛螺菌科共有序列进行比较。
79株毛螺菌科分离株与107株RDP毛螺菌科分离株的16S rRNA最大似然树。
结论
1.利用66种培养条件与16S rRNA基因测序相结合,允许培养粪便样本中平均95%丰度大于 0.1%的OTU。未培养的OTU在粪便中丰度较低。
2.CD与CI的测序进行比较表明,大多数OTU只能通过培养物检测到,突出了培养物在研究肠道微生物群多样性方面的优势。
3.将富含培养物的分子谱分析应用于目标毛螺菌科菌株,结果回收了79株菌株,其中12株在人类微生物组计划的“头号通缉”名单上。
微基生物推出的:微生物组学&培养组学联合检测套餐,大幅拓展可检测微生物的多样性。欢迎感兴趣朋友前来咨询了解。联系电话:021-50763698。

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用