微生物与肿瘤细胞的相互作用Unexpected guests in the tumor microenvironment: microbiome in cancer
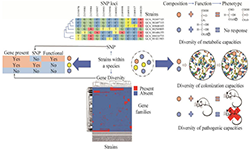
服务简介 具有复杂功能的微生物被发现是肿瘤微环境中的一个潜在成分。由于其生物量低和其他障碍,对内部微生物群的了解很少。微基生物对于肿瘤微生物的检测采用5R 16S测序的方法,大幅提高了细菌物种检测的覆盖率和分辨率。
5R 16S测序原理
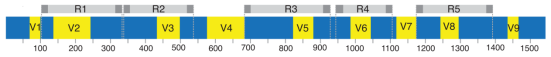
5R 16S扩增16S rRNA基因的5个区域 灰色R1-R5表示5R 16S扩增的5个区域
The human tumor microbiome is composed of tumor type–specific intracellular bacteria
对于肿瘤样本微生物的检测采用5R 16S测序,即对16S rRNA基因上的五个区域(V2、V3、V5、V6、V8)进行多重PCR扩增和测序。这种方法能兼容有一定程度降解的FFPE样本,并增加分析的分辨率。与V3 V4区扩增策略相比,扩增出的区域覆盖约68%的16S全长序列,可以大幅提高细菌物种检测的覆盖率和分辨率,尤其适合于低生物量的微生物样本检测。
肿瘤微生物组(Tumor Microbiome)或称瘤内微生物组(Intratumoral Microbiome)是肿瘤微环境中不可或缺的成员,与肿瘤的发生发展乃至治疗紧密相关。通过比较肿瘤内微生物群与体内其他部位的微生物群组成可能有助于我们识别存在于不同肿瘤中的关键微生物,为癌症预防提供新的见解。
目前16S rRNA基因扩增子测序和宏基因组学是鉴定肿瘤内微生物群最常用的方法。但由于肿瘤微生物生物量很低,肿瘤样本呈现出很高的宿主与细菌DNA比率,导致基于扩增子策略的16S rRNA基因测序,受宿主DNA干扰,引起菌群序列PCR扩增效率降低,甚至扩增失败。肿瘤微生物的全基因组测序则需要很好的微生物富集策略,否则可能无法进行。
基于以上情况,对于肿瘤样本微生物的检测采用5R 16S测序。
检测样本类型
肿瘤组织、FFPE等低载量生物样本
切片厚度20μm,10 张。
如果组织块(切片的一面)暴露在空气中时间比较久了,表面的DNA会容易降解,头三张片子不要。
如果病理组织很小块,片子量20张为宜。
相关文献研究
一、人类肿瘤微生物的分布 5R 16S rDNA测序方法

引言:细菌是人类肿瘤中众所周知的居民,但它们的存在是否对肿瘤有利或对细菌本身有利一直不清楚。研究人员对肿瘤微生物组进行了全面分析,研究了七种癌症类型的1526个肿瘤及其邻近的正常组织,包括乳腺癌、肺癌、卵巢癌、胰腺癌、黑色素瘤、骨癌和脑癌。
我们发现每种肿瘤类型都有不同的微生物组组成,而乳腺癌具有特别丰富多样的微生物组。瘤内细菌主要存在于细胞内,存在于癌细胞和免疫细胞中。我们还注意到肿瘤内细菌或其预测功能与肿瘤类型和亚型、患者的吸烟状况以及对免疫疗法的反应之间的相关性。
结果展示:1.细菌DNA、RNA和脂多糖存在于许多人类实体肿瘤中
研究7种实体肿瘤,它们代表了常见的癌症类型,而这些癌症的微生物群在很大程度上是未知的,如黑色素瘤、骨肿瘤和脑肿瘤。为了解决实验室携带的污染物,我们引入了643个与样本一起处理的阴性对照,其中包括437个DNA提取对照和206个PCR反应无模板对照。为解决样本到达实验室前可能发生的污染,我们还纳入了从研究中使用的石蜡块(无组织)边缘采集的168个纯石蜡样品。
我们分析了1010个肿瘤样本和516个正常样本,包括来自同一患病的正常邻近组织(NAT)。
发现,不同的癌症类型在对细菌DNA阳性的肿瘤比例上有所不同,从仅14.3%的黑色素瘤到60%的乳腺癌、胰腺癌和骨肿瘤。在与外部环境没有直接联系的实体肿瘤中也检测到了细菌DNA,
例如卵巢癌、多形性胶质母细胞瘤(GBM)和骨癌。
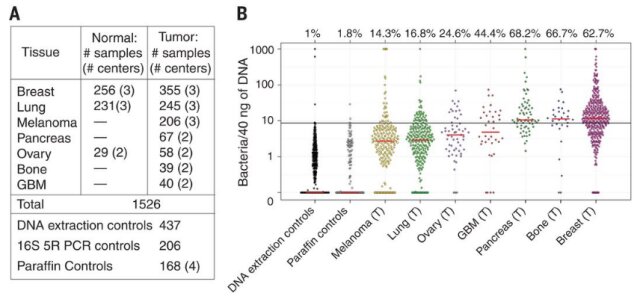
A研究中分析的人体样本数 B通过细菌16S rDNA qPCR评估人类肿瘤中细菌DNA的存在。
2. 瘤内细菌主要存在于细胞内,存在于癌细胞和免疫细胞中
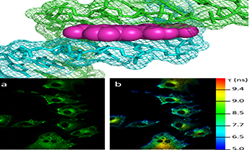
病理检查发现LPS和细菌16S rRNA主要分布于癌细胞和免疫细胞(图A)。在癌细胞中,细菌16S rRNA主要在细胞质中检测到,而LPS染色与细胞质和细胞核有关( 图B)。CD45阳性白细胞通过16SrRNA染色表现出比癌细胞更强的胞质细菌染色(图C)。LTA阳性细菌几乎全部存在于巨噬细胞中,通过免疫荧光在CD68中进行验证(图D)。LTA很少在癌细胞或CD45+/CD68−免疫细胞中检测到。虽然在CD45+/CD68+细胞中,细菌LPS和LTA染色强度非常强,但FISH在巨噬细胞中很少发现细菌16SrRNA(图A、D)。这种差异可能反映了巨噬细胞摄取了细菌成分,而不是活的细菌,也可能是由于在细菌被巨噬细胞吞噬和处理很长时间后,LPS和LTA在巨噬细胞中积累所致。此前已有研究表明,巨噬细胞对细菌脂多糖的处理非常缓慢;因此,在细菌被摄入和处理几个月后,可以在这些细胞中发现LPS。
为了进一步验证癌细胞内细菌的存在,我们对4例细菌LPS和16S rRNA阳性的人类乳腺肿瘤进行了相关光镜和电子显微镜(CLEM).结合LPS荧光染色和透射电镜(TEM)成像,清楚地显示了所有四种肿瘤中细菌的细胞内定位(图E)。
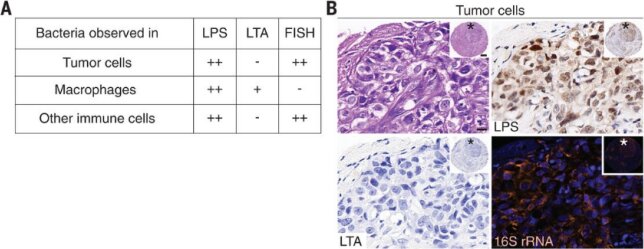
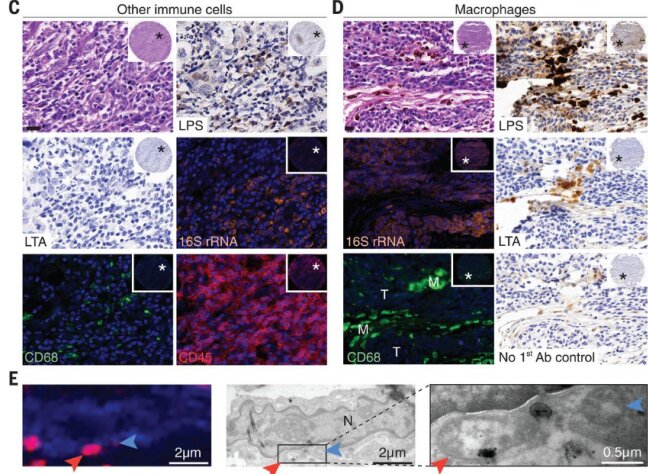
肿瘤内细菌存在于癌症细胞和免疫细胞中。
(A)在459、427和354个肿瘤核心中不同细胞类型中LPS、LTA和细菌16S rRNA的染色模式总结。CD45+/CD68+细胞被称为巨噬细胞;CD45+/CD68−细胞被称为其他免疫细胞。(B)细菌性LPS和16S rRNA在乳腺癌细胞中出现。(C)细菌性LPS和16S rRNA在一个高度炎症的乳腺肿瘤的CD45+/CD68−细胞中被证实。(D) A型黑色素瘤肿瘤显示典型的巨噬细胞相关菌(M)染色,LPS和LTA染色阳性,但无16S rRNA染色。附近的肿瘤细胞(T)显示典型的LPS和16S rRNA染色,LTA染色阴性。每个插图都显示了整个核心的低放大倍数。高倍率图像中的比例尺,20毫米。(E) CLEM显示了人类乳腺癌中的细胞内细菌。IF图像显示蓝色为DAPI,红色为LPS。两种细菌用箭头标记。
3.乳腺肿瘤的微生物组比其他肿瘤类型更丰富、更多样化
为了表征肿瘤内微生物组,我们开发了一种多重16S rDNA测序方案,可扩增16S rRNA基因上的五个短区域:5R 16S rDNA测序方法。通过使用短扩增子扩增68%的细菌16S rRNA基因,与广泛使用的V4或V3-V4扩增相比,该方法提高了细菌物种检测的覆盖度和分辨率。此外,它还可应用于源自福尔马林固定石蜡包埋肿瘤的部分降解DNA。
多重16S rDNA测序方案,在不同的肿瘤或正常组织中检测到9190种细菌。与其他所有待测肿瘤类型相比,乳腺肿瘤富含微生物组的多样性更高,在任何单个乳腺肿瘤样本中平均检测到16.4种细菌,而在所有其他肿瘤类型中平均<9种。另外,乳腺肿瘤样本中的细菌负荷和丰富度高于健康受试者正常乳腺样本中的细菌负荷和丰富度。
同时利用5名乳腺癌患者手术所得到的活体肿瘤组织,发现乳腺肿瘤中有来自三个主要门(变形菌门、厚壁菌门、放线菌门)的活细菌蛋白质,验证了人类肿瘤中存在活细菌。
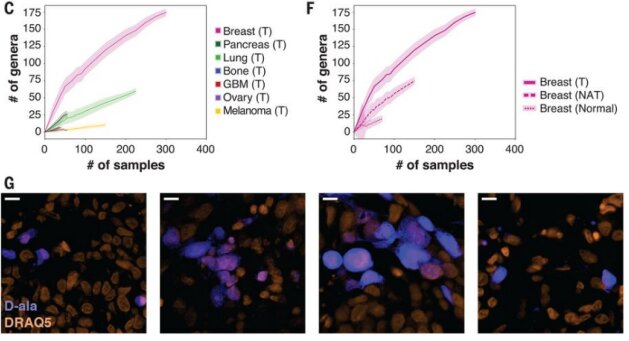
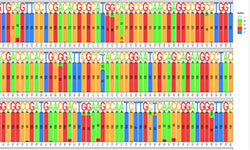
(A)细菌16SrRNA基因及其保守(蓝色)和可变(黄色)区域的图示。以大肠杆菌K-12亚株MG1655的序列为参考序列。多重5R PCR方法的5个扩增子用灰色表示。(B)应用于16S rDNA测序数据的分析管道的示意图。(C)稀疏图显示了每个选择的肿瘤样本数量中不同肿瘤类型中通过所有过滤器的细菌属的数量。所有样本香农多样性指数的(D) Box blot,按肿瘤类型分开。(E)每个肿瘤中存在细菌种类数量的Box blot检测。(F)在乳腺肿瘤、乳腺NAT和乳腺正常样本中通过所有过滤器的细菌属数量的稀疏性图。(G)用荧光标记的d-丙氨酸在体外培养2小时的四种人乳腺肿瘤的荧光图像(蓝色)。
4.不同的肿瘤类型具有不同的微生物组成
使用单一测序方法和平台来表征多种肿瘤类型中的微生物组,使我们能够直接比较这些肿瘤的微生物组。比较某一特定肿瘤类型内和不同肿瘤类型内所有样本对之间的β多样性,发现同一类型肿瘤的微生物组往往比其他肿瘤类型的微生物组更相似。同时,肿瘤类型独特的微生物组组成在物种水平上也很明显。
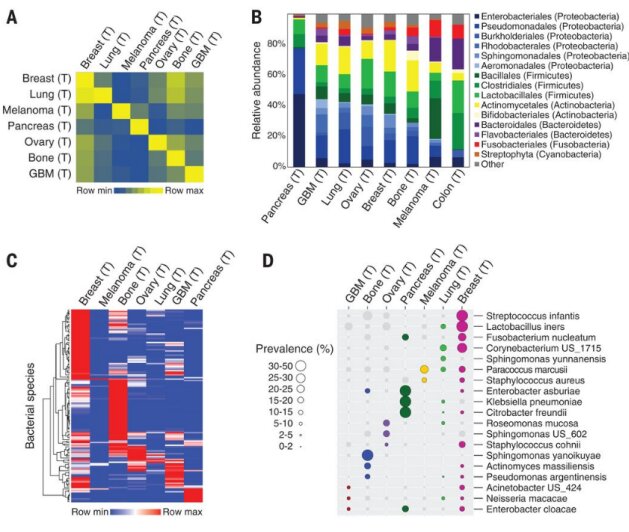
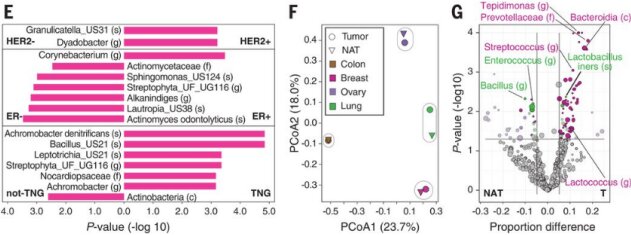
(A)Jaccard相似性指数是根据所有可能的样本对之间通过肿瘤(n=528)中所有过滤器的细菌种类概况计算的。热图显示了来自任何两种癌症类型的样本对之间所有指数的平均值。(B)不同肿瘤类型的有序级系统发育型分布。(C)137种细菌流行率的无监督层次聚类,这些细菌在一种肿瘤类型中被击中,并且在至少一种肿瘤类型中存在于10%或更多的样本中。(D)来自(C)的19种细菌的流行,显示在不同的肿瘤类型中。(E)不同乳腺肿瘤亚型之间流行率存在显着差异的细菌类群显示在条形图中。(F)主坐标分析(PCoA)双标图在不同组织类型的细菌物种谱之间的Jaccard相似性指数。(G)火山图显示了乳房、肺和卵巢样本中肿瘤(T)及其NAT之间细菌的差异流行。
在本研究中,我们对来自7种人类肿瘤类型的1526个样本的微生物组进行了特征分析。采取了多种措施来最小化和控制污染,并使用5R多重细菌16S rDNA PCR测序技术来获得物种水平的分辨率。通过使用单一平台探索多种肿瘤类型,可以比较不同的肿瘤类型,并发现癌症类型特异性的微生物特征。
二、肿瘤中微生物的认识

随着对宿主-微生物相互作用的深入研究,肿瘤组织中存在的瘤内微生物群的概念被提出。肿瘤内微生物在19世纪首次被观察到并被描述,但在接下来的一个世纪中该领域几乎没有取得任何进展。近年来,随着检测技术的发展和对肿瘤微环境(TMEs)的深入了解,越来越多的证据证实了肿瘤内细菌的存在。
瘤内微生物群的来源
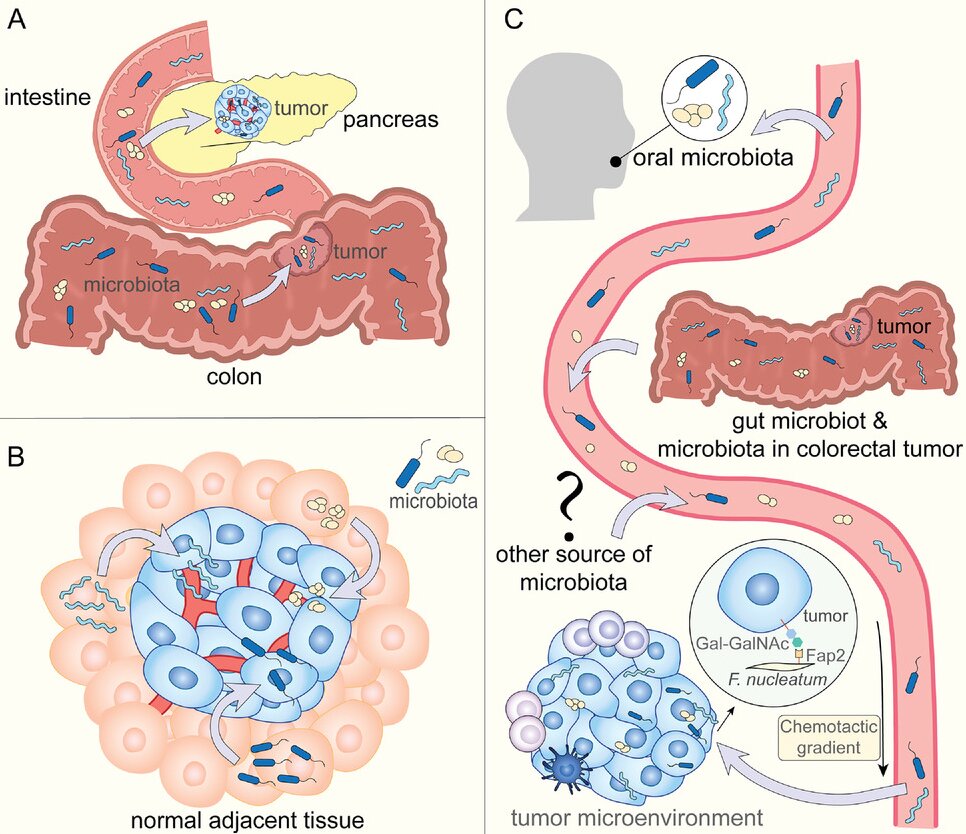
肿瘤内微生物群的来源:A) 粘膜器官。肠道微生物会扰乱粘膜屏障并进入肿瘤部位,而胰腺癌的瘤内细菌则通过胰管进入肿瘤部位。B) NAT是肿瘤内微生物群的潜在来源。C) 循环系统。肿瘤内微生物通过血行传播从口腔、肠道、肿瘤和其他部位进入肿瘤部位。
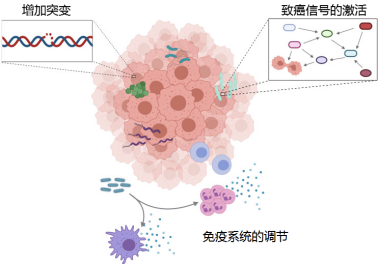
肿瘤内微生物群影响肿瘤发生和治疗的机制
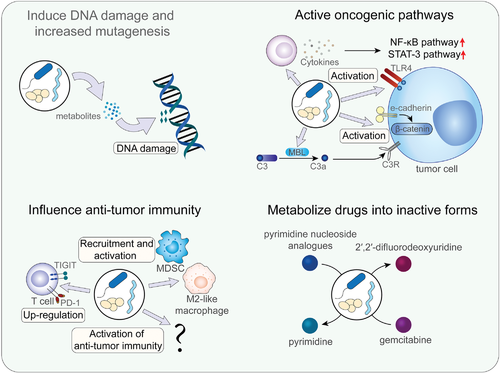
诱导肿瘤发生的两个主要机制包括引起DNA损伤和激活致癌途径。肿瘤内微生物群也影响抗肿瘤免疫并发挥复杂的作用。此外,肿瘤内微生物群代谢化疗药物并导致化疗耐药。
不同肿瘤的瘤内微生物群的异质性
TME具有高度异质性,在不同的癌症中,瘤内微生物的组成和丰度也存在显着差异。因此,肿瘤内微生物群对不同癌症的影响也可能不同。
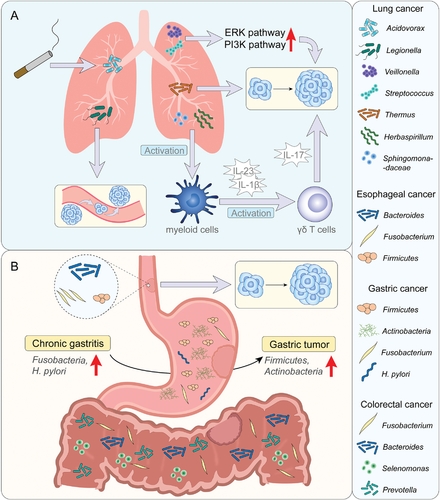
不同肿瘤中肿瘤内微生物群的异质性 A) 肺肿瘤。有几种细菌会影响肺癌的进展和转移。这些细菌通过不同的途径发挥作用。
B)胃肠道肿瘤。胃肠道肿瘤中微生物群的组成和功能很复杂。厚壁菌门、硒单胞菌门等多种细菌与肿瘤的进展密切相关。
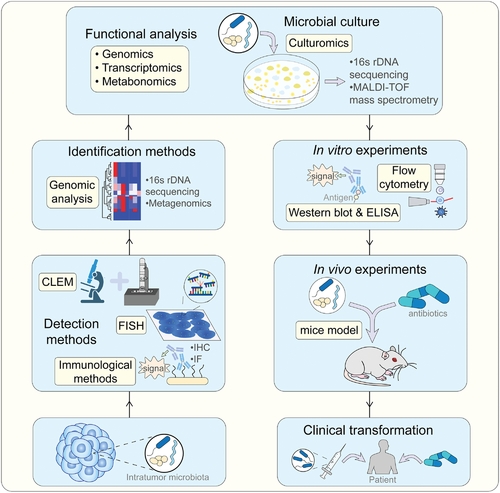
研究肿瘤内微生物群的方法
肿瘤内微生物群和抗肿瘤治疗
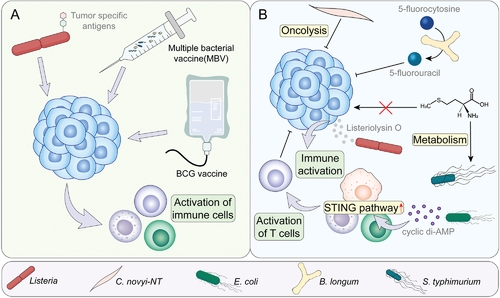
肿瘤内微生物群是TME的重要且异质的组成部分,在肿瘤的发生和发展中发挥着复杂的作用。该领域还有很多问题有待解决,需要技术的发展和努力。值得注意的是,肿瘤内微生物群的研究具有强大的临床转化潜力,可能成为抗肿瘤治疗的下一个热点。
三、低微生物生物量微生物组研究中的污染:问题和建议

本研究首先回顾了污染物DNA和交叉污染是如何在微生物组研究中产生的,并讨论了它们的负面影响,特别是在低微生物生物量样本的分析期间。然后,确定了研究人员可以采取的几个关键措施,以减少污染DNA和交叉污染的影响。
DNA污染影响微生物组研究
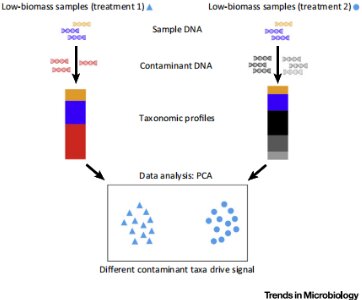
低微生物生物量样品的两个处理组(三角形和圆形)在微生物组成上没有差异(样品DNA颜色相同,蓝色和橙色)。然而,由于处理组处理背景不同的,污染物DNA的类型和丰度的差异(红色和黑色)驱动了信号,从而导致处理组有不同的微生物组成。
减少DNA污染采取的措施
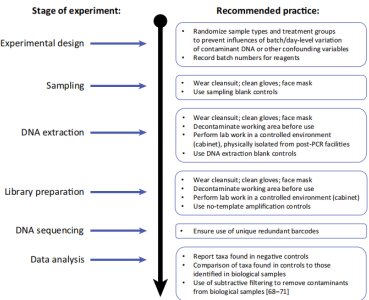
在低微生物生物量中,减少实验偏差和引入污染物DNA的措施。
肿瘤微生物组的生物量相对较低,宿主DNA和环境微生物DNA的污染是最大的障碍。
微基生物对于肿瘤微生物等低微生物样本的检测采用5R 16S测序的方法,大幅提高了细菌物种检测的覆盖率和分辨率。欢迎前来咨询了解:021-50763698。

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用